Омега-петля
Петля омега — це нерегулярний структурний мотив білка[1][2], що складається з петлі з шести і більше амінокислотних залишків і будь-якої послідовності амінокислот. Визначальною характеристикою є те, що залишки, що складають початок і кінець петлі, близькі між собою в просторі, не мають втручаються довжин звичайних вторинних структурних мотивів. Він названий за своєю формою, що нагадує велику грецьку букву Омега (Ω).
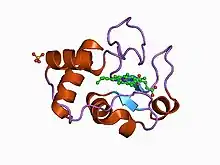
Структура
Петлі Омеги, будучи нерегулярними, неповторюваними вторинними структурними одиницями, мають різноманітні тривимірні форми. Форми петлі Омеги аналізуються для виявлення повторюваних візерунків у двогранних кутах та загальної форми петлі, щоб допомогти визначити потенційну роль у складі та функціонуванні білка[3][4].
Оскільки петлі майже завжди знаходяться на поверхні білка, часто передбачається, що ці структури є гнучкими; однак, різні омега-петлі виявляють діапазон гнучкості в різні часові шкали руху білка і були визначені як ті, що відіграють роль у згортань деяких білків, включаючи зворотну транскриптазу ВІЛ-1; цитохром c; і нуклеїди.[5][6]
Примітки
- Leszczynski, J. F.; Rose, G. D. (14 листопада 1986). Loops in globular proteins: a novel category of secondary structure. Science (англ.) 234 (4778). с. 849–855. ISSN 0036-8075. PMID 3775366. doi:10.1126/science.3775366. Процитовано 22 грудня 2019.
- Fetrow, J S (1 червня 1995). Omega loops: nonregular secondary structures significant in protein function and stability.. The FASEB Journal 9 (9). с. 708–717. ISSN 0892-6638. doi:10.1096/fasebj.9.9.7601335. Процитовано 22 грудня 2019.
- Pal, Manoj; Dasgupta, Swagata (2003). The nature of the turn in omega loops of proteins. Proteins: Structure, Function, and Bioinformatics (англ.) 51 (4). с. 591–606. ISSN 1097-0134. doi:10.1002/prot.10376. Процитовано 22 грудня 2019.
- Dhar, Jesmita; Chakrabarti, Pinak (1 червня 2015). Defining the loop structures in proteins based on composite β-turn mimics. Protein Engineering, Design and Selection (англ.) 28 (6). с. 153–161. ISSN 1741-0126. doi:10.1093/protein/gzv017. Процитовано 22 грудня 2019.
- Mager, P. P. (1996-12). Molecular simulation of the folding patterns of the omega-loop (Tyr181 to Tyr188) of HIV-1 reverse transcriptase. Drug Design and Discovery 14 (3). с. 213–223. ISSN 1055-9612. PMID 9017364. Процитовано 22 грудня 2019.
- Maity, Haripada; Rumbley, Jon N.; Engl, S. Walter. Functional Role of a Protein Foldon—An-Loop Foldon Controls the Alkaline Transition in Ferricytochrome c.