Складання геному
Складання геному — процес об'єднання великої кількості коротких фрагментів ДНК (рідів) у одну або кілька довгих послідовностей (контигів і скаффолдів) з метою відновлення послідовностей ДНК хромосом, з яких виникли ці фрагменти в процесі секвенування.
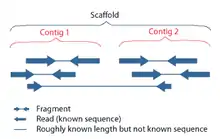
Складання геному є дуже складною обчислювальною задачею, зокрема, ускладненої тим, що геноми часто містять багато однакових повторюваних послідовностей (так звані геномні повтори). Ці повтори можуть мати довжину кілька тисяч нуклеотидів, а також зустрічатися у геномі в тисячі різних місць. Особливо багаті повторами великі геноми рослин і тварин, зокрема й геном людини.
Алгоритмічні підходи
Існує два підходи для складання геномів — заснований на перекритті overlap-layout-consensus (застосовується для довгих фрагментів), а також заснований на графах де Брейна (застосовується для коротких фрагментів)[1][2].
Overlap-Layout-Consensus
При секвенуванні методом дробовика всі ДНК організму спочатку розрізають на мільйони маленьких фрагментів довжиною до 1000 нуклеотидів. Потім алгоритми складання геному розглядають отримані фрагменти одночасно, знаходячи їх перекриття (overlap), об'єднуючи їх за перекриттями (layout) і виправляючи помилки в об'єднаному рядку (consensus). Ці кроки в процесі складання можуть повторюватися кілька разів.
Цей підхід до складання геномів був найпоширенішим до появи методів секвенування наступного покоління.
Графи де Брейна
З розвитком технологій секвенування наступного покоління отримання фрагментів стало на порядок дешевшим, але розмір фрагментів став меншим (до 150 нуклеотидів), а кількість помилок при читанні фрагментів зросла (до 3 %). При складанні таких даних набули поширення методи[3], засновані на графах де Брейна.
Доступні складальники
Список популярних геномових складальників:
| Назва | Підтримувані технології | Автори | Представлений | Оновлений | Ліцензія* | Домашня сторінка |
|---|---|---|---|---|---|---|
| ABySS | Solexa, SOLiD | Simpson, J. та інші | 2008 | 2011 | NC-A | посилання |
| ALLPATHS-LG | Solexa, SOLiD | Gnerre, S. et та інші | 2011 | 2011 | OS | посилання |
| CLC Genomics Workbench | Sanger, 454, Solexa, SOLiD | CLC bio | 2008 | 2010 | C | посилання |
| Euler | Sanger, 454 (, Solexa ?) | Pevzner, P. та інші | 2001 | 2006 | (C/NC-A?) | посилання |
| Euler-sr | 454, Solexa | Chaisson, MJ. та інші | 2008 | 2008 | NC-A | посилання |
| IDBA | Sanger, 454, Solexa | Yu Peng, Henry CM Leung, Siu-Ming Yiu, Francis YL Chin | 2010 | 2010 | (C/NC-A?) | посилання |
| MIRA | Sanger, 454, Solexa | Chevreux, B. | 1998 | 2011 | OS | посилання |
| Newbler | 454, Sanger | 454 / Roche | 2009 | 2009 | C | посилання |
| SOPRA | Illumina, SOLiD, Sanger, 454 | Dayarian, A. та інші | 2010 | 2011 | OS | посилання |
| SOAPdenovo | Solexa | Li, R. та інші | 2009 | 2009 | OS | посилання |
| SPAdes | Illumina, Solexa | Bankevich, A та інші | 2012 | 2012 | OS | посилання |
| Velvet | Sanger, 454, Solexa, SOLiD | Zerbino, D. та інші | 2007 | 2009 | OS | посилання |
| Canu | PacBio, Oxford Nanopore | Koren, S. та інші | 2017 | 2020 | OS | посилання |
| * Ліцензії: OS = Open Source; C = комерційна; C/NC-A = комерційна, але безплатна для використання в некомерційних і наукових цілях; в дужках = невідомо, але найпевніше C/NC-A | ||||||
Примітки
- Zhenyu Li et al. Comparison of the two major classes of assembly algorithms: overlap–layout–consensus and de-bruijn-graph // Briefings in Functional Genomics : journal. — 2012. — Vol. 11, no. 1 (23 June). — P. 25—37. — DOI:.
- Miller J. R., Koren S., Sutton G. Assembly algorithms for next-generation sequencing data // Genomics : journal. — Academic Press, 2010. — Vol. 95, no. 6 (23 June). — P. 315—327.
- Pavel A. Pevzner, Haixu Tang, Michael S. Waterman. An Eulerian path approach to DNA fragment assembly // Proceedings of the National Academy of Sciences of the United States of America : journal. — 2001. — Vol. 98, no. 17 (23 June). — P. 9748—9753. — DOI:.