КАСП (експеримент)
КАСП (англ. CASP) — всесвітній експеримент з Критичного Аналізу методів для Структурних Передбачень білків. КАСП є «полігоном» для тестування методів з передбачення структури білків, знаряддям для незалежного і неупередженого аналізу прогресу і проблем у галузі комп'ютерного моделювання білків, і рушійною силою для розробки нових і покращення існуючих методів моделювання. Хоча основною метою КАСПу є аналіз поточного стану справ в області моделювання структури білків, багато хто розглядає експеримент як першість світу у цій галузі науки. Понад 100 наукових осередків зі всього світу регулярно беруть участь у КАСПі, і не є нетиповою ситуація, коли цілі наукові колективи відкладають свої інші дослідницькі проекти на кілька місяців з тим, щоб сконцентруватись на підготовці своїх серверів до «змагання» і займатись безпосередньо моделюванням запропонованих білків.
Організація експерименту і його загальні принципи
КАСП було започатковано Джоном Молтом (John Moult, Мерилендський Університет) і Кшиштофом Фіделісом (Krzysztof Fidelis, Ліверморська національна лабораторія) 1994 року[1]. Починаючи з 2004 року одним з організаторів експерименту є українець Андрій Криштафович (Andriy Kryshtafovych, Каліфорнійський університет в Девісі). Організатори відповідають за всі аспекти проведення експерименту, зокрема за достарчання послідовностей білків для моделювання, дотримання конфіденційності під час експерименту, організацію оцінювання передбачень і проведення конференції моделерів для обговорення результатів чергового туру передбачень. Незалежні експерти [недоступне посилання з липня 2019] запрошуються організаторами для оцінки моделей і складання висновків про об'єктивний стан справ в галузі. Участь в експерименті може взяти кожен охочий, зареєструвавши дослідницьку групу.
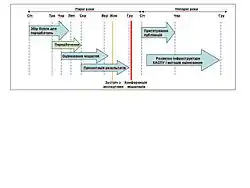
Експеримент проводиться раз в два роки. Головна суть КАСПу полягає в тому, що моделерам пропонуються для передбачення білки (на КАСПівському сленгу — «мішені»), структури яких ще невідомі на час моделювання. Це є зазвичай пептидні послідовності, що невдовзі будуть розв'язані експериментально за допомогою рентгенівської кристалографії чи ЯМР-спектроскопії, або структури, що були щойно розв'язані (частіше всього одним з центрів структурної геноміки), але ще не доступні публічно зі структурної бази даних білків PDB . У час, відведений для передбачень, дослідницькі групи повинні згенерувати модель і надіслати її до Центру передбачень білків . У центрі[2] моделі автоматично оцінюються за допомогою стандартних процедур[3], після чого результати обчислень передаються незалежним експертам, які інтерпретують і підсумовують ці дані, а також аналізують деталі моделей за допомогою додаткових методів. Авторство моделей розкривається для експертів-оцінювальників лише після того, як вони складуть звіт організаторам про якість моделей. Після цього найкращі групи запрошуються на конференцією моделерів, яка завершує цикл КАСПу, і де обговорюються результати останнього експерименту і пропонуються зміни до наступного. Принципи «сліпого» передбачення і оцінки забезпечують об'єктивність експерименту, дозволяють викривати переваги чи вади окремих методів, ставлять бар'єр для непідтверджених заявок про успіх і надають розробникам методів механізм для встановлення їхньої дійсної спроможності.
Основні типи методів для моделювання структур
Методи для передбачення третинної структури білків оцінюються в КАСПі окремо для шаблонного і безшаблонного моделювання. Якщо запропоновані для передбачення білки мають еволюційну схожість до білків з відомою структурою — так званого шаблону (англ. — template) — то тоді для моделювання структури білка застосовують принцип моделювання за шаблоном (англ. — template-based modeling або comparative modeling). Коли не вдається знайти шаблону, то тоді застосовуються так звані безшаблонні методи (англ. template-free methods або de novo methods), які зазвичай використовують техніку складання білків з коротких сегментів відомих структур або наближені стратегії пошуку мінімуму енергетичного ландшафту. Із зростом бази даних структур стає все важче відшукати для експерименту білки, придатні для тестування методів безшаблонного моделювання[4].
Оцінка моделей
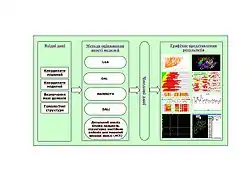
У КАСПі оцінюються передбачення в таких категоріях:
- передбачення третинної структури білків (результати шаблонного і безшаблонного моделювання оцінюються окреио)
- передбачення вторинної структури (КАСП1-КАСП5)
- структурні комплекси (КАСП2; окремий експеримент — КАПРІ (англ. — CAPRI) — продовжує цю тематику)
- передбачення контактів між амінокислотними залишками (починаючи з КАСП4)
- передбачення невпорядкованих регіонів (починаючи з КАСП5)
- передбачення границь доменів (КАСП6-КАСП8)
- передбачення функції (починаючи з КАСП6)
- оцінка якості моделей (починаючи з КАСП7)
- покрашення моделей (починаючи з КАСП7)
- шаблонні передбачення високої точності (починаючи з КАСП7).
Основним методом оцінки якості структурних моделей в КАСПі є зіставлення позицій α-вуглецевих атомів в моделі і мішені. Це порівняння проводиться за допомогою пакету програм[5], базовою з яких є LGA[6]. Основною функцією оцінки якості моделі є міра GDT_TS (з програми LGA), що описує процент вірно змодельованих амінокислотних залишків в моделі по відношенню до мішені. Результати аналізу наводяться на графіках, що підсумовують відстані між парами еквівалентних α-вуглеців в оптимальному накладенні моделі на експериментальну структуру. Слід зауважити проте, що результати автоматичного числового аналізу є ненадійними у випадках, коли моделі є досить далекими від нативних структур, що звикло трапляється при безшаблонному моделюванні. У цих випадках основним механізмом оцінки якості моделей є їхній візуальний аналіз експертами[7]. Шаблонні передбачення високої точності оцінювалися в КАСП7 на предмет їхньої придатності для фазування молекулярного заміщення кристалічної структури мішені[8], а в КАСП8 — на предмет відповідності повно-атомних моделей (а не лише α-вуглецевих) експериментальним структурам[9].
Публікація результатів
Результати експерименту друкуються в спеціальному номері наукового журналу Proteins: Structure, Function, Bioinformatics. Посилання до всіх КАСПових примірників журналу наведені на вебсайті Центру передбачень . Заголовна стаття кожного спеціального випуску описує специфіку кожного з експериментів[10], а завершальна стаття наводить аналіз прогресу в області моделювання[11],[12].
Результати ранкування методів
Офіційні результати останніх двох КАСПів опубліковані організаторами експерименту на вебсайті Центру передбачень білків:
- КАСП9 — Офіційний ранк автоматичних серверів
- КАСП9 — Офіційний ранк всіх методів
- КАСП8 — Офіційний ранк автоматичних серверів
- КАСП8 — Офіційний ранк всіх методів
Зауважимо, що не всі сервери, що беруть участь в КАСПі, є публічно доступними після завершення експерименту. Також, багато публічних серверів з найвищими КАСПовими ранками, мають довгі черги на моделювання, і тому інколи потрібно довгий час, щоб отримати передбачення.
Кілька дослідників також зробили свої незалежні ранкування методів з КАСП8 (2008) і опублікували їх на Інтернеті. Наприклад:
Див. також
Посилання
- Moult, J., et al. (1995). A large-scale experiment to assess protein structure prediction methods. Proteins: Structure, Function, Genetics 23 (3): ii–iv.
- Kryshtafovych, A., et al. (2009). Protein structure prediction center in CASP8. Proteins: Structure, Function, Bioinformatics 77 (S9): 5–9.
- Cozzetto, D., et al. (2009). Evaluation of template-based models in CASP8 with standard measures. Proteins: Structure, Function, Bioinformatics 77 (S9): 18–28.
- Tress, M., et al. (2009). Target domain definition and classification in CASP8. Proteins: Structure, Function, Bioinformatics 77 (S9): 10–17.
- Kryshtafovych, A., et al. (2007). New tools and expanded data analysis capabilities at the protein structure prediction center. Proteins: Structure, Function, Bioinformatics 69 (S8): 19–26.
- Zemla, A. (2003). LGA: a method for finding 3D similarities in protein structures. Nucleic Acids Research 31: 3370–3374.
- Ben-David, M., et al. (2009). Assessment of CASP8 structure predictions for template free targets. Proteins: Structure, Function, Bioinformatics 77 (S9): 50–65.
- Read, R.J., Chavali, G. (2007). Assessment of CASP7 predictions in the high accuracy template-based modeling category. Proteins: Structure, Function, Bioinformatics 69 (S8): 27–37.
- Keedy, D.A. (2009). The other 90% of the protein: Assessment beyond the α-carbon for CASP8 template-based and high-accuracy models. Proteins: Structure, Function, Bioinformatics 77 (S9): 50–65.
- Moult, J., et al. (2009). Critical assessment of methods of protein structure prediction (CASP) - round VIII. Proteins 77 (S9): 1–4.
- Kryshtafovych, A., et al. (2005). Progress over the first decade of CASP experiments. Proteins: Structure, Function, Bioinformatics 61 (S7): 225–236.
- Kryshtafovych, A., et al. (2009). CASP8 results in context of previous experiments. Proteins: Structure, Function, Bioinformatics 77 (S9): 217–228.