Спіраль-поворот-спіраль
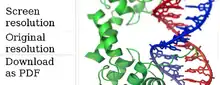
У білках спіраль-поворот-спіраль (HTH) є головним структурним мотивом, здатним зв'язувати ДНК. Кожен мономер містить два спіралі α, з'єднані короткою ланцюгом амінокислот, які зв'язуються з основною канавкою ДНК. Мотив HTH зустрічається у багатьох білках, які регулюють експресію генів. Його не слід плутати з мотивом helix-loop-helix.[1]

Відкриття
Відкриття мотиву helix-turn-helix базувалося на подібності між декількома генами, що кодують регуляторні білки транскрипції з бактеріафага лямбда та кишкової палички: Cro, CAP та репресора λ, для яких було встановлено спільну 20-25 амінокислотну послідовність, полегшує розпізнавання ДНК.[2][3]
Функція
Мотив helix-turn-helix — це мотив, що зв'язує ДНК. Розпізнавання і зв'язування з ДНК білками helix-turn-helix здійснюється двома α-спіралями, один займає N-кінцевий кінець мотиву, а другий на C-кінці. У більшості випадків, наприклад, у репресора Cro, друга спіраль найбільше сприяє розпізнаванню ДНК, і тому її часто називають «спіраллю розпізнавання». Він пов'язується з основною канавкою ДНК через низку водневих зв'язків та різних взаємодій Ван-дер-Ваальса з відкритими базами. Інша спіраль α стабілізує взаємодію білка і ДНК, але не відіграє особливо сильної ролі в його розпізнаванні. Спіраль розпізнавання та її попередня спіраль завжди мають однакову відносну орієнтацію.[2]
Класифікація
Було зроблено кілька спроб класифікації мотивів спіраль-поворот-спіраль на основі їх структури та просторового розташування їх спіралей. Деякі основні типи описані нижче.[3]
Ді-спіраль
Мотив ді-спіраль-спіраль-поворот-спіраль — це найпростіший мотив helix-turn-helix. Фрагмент вбудованого гомеодомену, що охоплює лише дві спіралі і надзвичайно швидко утворюється незалежно від складності білка.[4]
Триспіральний
Приклад цього мотиву знаходимо в активаторі транскрипції Myb.[5]
Тетра-спіральний
Мотив тетра-спіральної спіралі-повороту-спіралі має додаткову С-кінцеву спіраль порівняно з три-спіральними мотивами. До них належать ДНК-зв'язуючий домен HTH типу LuxR, виявлений у факторах транскрипції бактерій, і мотив helix-turn-helix, знайдений у репресорах TetR. Також зустрічаються багатоспіральні версії з додатковими спіралями.
Крилатий спіраль-поворот-спіраль
Крилатий мотив крила-повороту-спіралі (wHTH) утворений 3-спіральним пучком та 3-х або 4-х нитковим бета-аркушем (крилом). Топологія спіралей і ниток в мотивах wHTH може відрізнятися.[6]
Інші модифіковані мотиви спіралі-повороту-спіралі
Інші похідні мотиву helix-turn-helix включають домен, що зв'язує ДНК, знайдений у MarR, регулятор множинної стійкості до антибіотиків, який утворює крилату спіраль-поворот-спіраль з додатковою спіраллю C-кінцевою альфа.
Примітки
- Brennan, R. G.; Matthews, B. W. (5 лютого 1989). The helix-turn-helix DNA binding motif. The Journal of Biological Chemistry 264 (4). с. 1903–1906. ISSN 0021-9258. PMID 2644244. Процитовано 22 грудня 2019.
- Matthews, B W; Ohlendorf, D H; Anderson, W F; Takeda, Y (1982-03). Structure of the DNA-binding region of lac repressor inferred from its homology with cro repressor.. Proceedings of the National Academy of Sciences of the United States of America 79 (5). с. 1428–1432. ISSN 0027-8424. PMID 6951187. Процитовано 22 грудня 2019.
- Pfam: Family: HTH_Tnp_1 (PF01527). pfam.xfam.org. Процитовано 22 грудня 2019.
- Religa, Tomasz L.; Johnson, Christopher M.; Vu, Dung M.; Brewer, Scott H.; Dyer, R. Brian; Fersht, Alan R. (29 травня 2007). The helix–turn–helix motif as an ultrafast independently folding domain: The pathway of folding of Engrailed homeodomain. Proceedings of the National Academy of Sciences of the United States of America 104 (22). с. 9272–9277. ISSN 0027-8424. PMC 1890484. PMID 17517666. doi:10.1073/pnas.0703434104. Процитовано 22 грудня 2019.
- Ogata, K; Hojo, H; Aimoto, S; Nakai, T; Nakamura, H; Sarai, A; Ishii, S; Nishimura, Y (15 липня 1992). Solution structure of a DNA-binding unit of Myb: a helix-turn-helix-related motif with conserved tryptophans forming a hydrophobic core.. Proceedings of the National Academy of Sciences of the United States of America 89 (14). с. 6428–6432. ISSN 0027-8424. PMID 1631139. Процитовано 22 грудня 2019.
- Donaldson, L W; Petersen, J M; Graves, B J; McIntosh, L P (2 січня 1996). Solution structure of the ETS domain from murine Ets-1: a winged helix-turn-helix DNA binding motif.. The EMBO Journal 15 (1). с. 125–134. ISSN 0261-4189. PMID 8598195. Процитовано 22 грудня 2019.