Слизька послідовність
Слизька́ послідо́вність (англ. slippery sequence) — це невелика частина кодонної нуклеотидної послідовності (зазвичай УУУАААС), яка контролює швидкість і шанс рибосомального зсуву рамки зчитування. Слизька послідовність спричиняє швидше рибосомальне пересування, що, у свою чергу, може спричинити «ковзання» зчитування рибосоми. Це дозволяє тРНК зсуватися на 1 основу (-1) після того, як вона спарується зі своїм антикодоном, змінюючи рамку зчитування[2][3][4][5][6]. −1 зсув рамки зчитування, запущений такою послідовністю, є запрограмованим рибосомальним зсувом рамки зчитування. За ним йде спейсерна область та вторинна структура РНК. Такі послідовності поширені у вірусних поліпротеїнах[1].
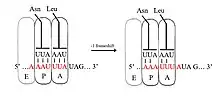
Зсув рамки зчитування відбувається через зміщене (воблівське) спаровування основ. Знижена вільна енергія Гіббза вторинних структур вказує на те, як часто відбувається зсув рамки зчитування[7]. Напруження на молекулі мРНК також відіграє певну роль[8]. Список слизьких послідовностей, виявлених у вірусів тварин, наведено у публікації Хуан та ін[9].
Слизькі послідовності, які спричиняють 2-основне «ковзання» (−2 зсув рамки зчитування), були побудовані з УУУУУУА-послідовності ВІЛ[8].
Посилання
- Jacks T, Madhani HD, Masiarz FR, Varmus HE (November 1988). Signals for ribosomal frameshifting in the Rous sarcoma virus gag-pol region. Cell 55 (3): 447–58. PMID 2846182. doi:10.1016/0092-8674(88)90031-1.
- Green L, Kim CH, Bustamante C, Tinoco I (January 2008). Characterization of the mechanical unfolding of RNA pseudoknots. Journal of Molecular Biology 375 (2): 511–28. PMID 18021801. doi:10.1016/j.jmb.2007.05.058.
- Yu CH, Noteborn MH, Olsthoorn RC (December 2010). Stimulation of ribosomal frameshifting by antisense LNA. Nucleic Acids Research 38 (22): 8277–83. PMC 3001050. PMID 20693527. doi:10.1093/nar/gkq650.
- Dr Ian Brierley Research description. Department of Pathology, University of Cambridge. Архів оригіналу за 2 жовтня 2013. Процитовано 28 липня 2013.
- Molecular Biology: Frameshifting occurs at slippery sequences. Molecularstudy.blogspot.com. 16 жовтня 2012. Процитовано 28 липня 2013.
- Farabaugh PJ, Björk GR (March 1999). How translational accuracy influences reading frame maintenance. The EMBO Journal 18 (6): 1427–34. PMC 1171232. PMID 10075915. doi:10.1093/emboj/18.6.1427.
- Cao S, Chen SJ (March 2008). Predicting ribosomal frameshifting efficiency. Physical Biology 5 (1): 016002. Bibcode:2008PhBio...5a6002C. PMC 2442619. PMID 18367782. doi:10.1088/1478-3975/5/1/016002.
- Lin Z, Gilbert RJ, Brierley I (September 2012). Spacer-length dependence of programmed -1 or -2 ribosomal frameshifting on a U6A heptamer supports a role for messenger RNA (mRNA) tension in frameshifting. Nucleic Acids Research 40 (17): 8674–89. PMC 3458567. PMID 22743270. doi:10.1093/nar/gks629.
- Huang X, Cheng Q, Du Z (2013). A genome-wide analysis of RNA pseudoknots that stimulate efficient -1 ribosomal frameshifting or readthrough in animal viruses. BioMed Research International 2013: 984028. PMC 3835772. PMID 24298557. doi:10.1155/2013/984028.