Cas9
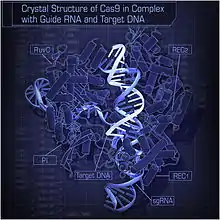
Cas9 — (англ. CRISPR associated protein 9, CRISPR-асоційований білок) — це керована за допомогою РНК-гідів ендонуклеаза, пов'язана з адаптивною імунною системою CRISPR (англ. Clustered Regularly Interspaced Palindromic Repeats) у ряду бактерій, зокрема Streptococcus pyogenes. S. pyogenes використовує Cas9 для запам'ятовування[2], подальшого розпізнавання та розрізання чужорідної ДНК[3], наприклад, ДНК бактеріофагів або плазмід.
| hypothetical protein SPy_1046 | |||||||
|---|---|---|---|---|---|---|---|
| Ідентифікатори | |||||||
| Символи | SPy_1046hypothetical proteinSPy1046 | ||||||
| Зовнішні ІД | GeneCards: | ||||||
| Ортологи | |||||||
| Види | Людина | Миша | |||||
| Entrez |
|
| |||||
| Ensembl |
|
| |||||
| UniProt |
|
| |||||
| RefSeq (мРНК) |
|
| |||||
| RefSeq (білок) |
|
| |||||
| Локус (UCSC) | н/д | н/д | |||||
| PubMed search | [1] | н/д | |||||
| Вікідані | |||||||
| |||||||
Cas9 розпізнає чужорідну ДНК розкручуючи її і визначає її комплементарність до двадцяти спарених основ спейсера керуючої РНК. Якщо субстрат комплементарний керуючій РНК, Cas9 розщеплює чужу ДНК. У цьому сенсі механізм CRISPR-Cas9 має ряд паралелей з механізмом РНК-інтерференції (RNAi) у еукаріот. Безпека практичного застосування даного методу визначається в тому числі і тим фактором, чи є шукана послідовність двадцяти спарених основ унікальною в ДНК, що модифікується.
Використання Cas9 в геннії інженерії
Крім своєї початкової ролі в бактеріальному імунітеті, білок Cas9 активно використовують для створення точкових розривів подвійної спіралі ДНК, такі розриви можуть призводити до інактивації генів або створення гетерологічних генів за допомогою з'єднання негомологічних кінців і відповідної гомологічної рекомбінації. Разом з білками ZFN і TALEN, Cas9 стає важливим інструментом в сфері редагування геному. Більш того, створена технологія, що дозволяє вносити точкову мутацію не розрізаючи ланцюг ДНК, а шляхом перетворення однієї нуклеотидної основи на іншу[4].
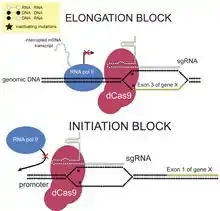
До 2012 року Cas9 отримав широку популярність, тому що він дозволяє розрізати практично будь-яку нуклеотидну послідовність, комплементарну керуючій РНК[3]. Оскільки вибірковість Cas9 базується на комплементарності керуючої РНК і ДНК, а не модифікації самого білка (на відміну від випадків TALEN і ZFN), для нових ДНК-мішеней можливе вироблення специфічних Cas9[5]. Варіації Cas9, які зв'язують ДНК але не розрізають її (dCas9), можуть бути використані для доставки транскрипційних активаторів або репресорів до специфічним послідовностей ДНК з метою регулювання транскрипційної активації і репресії[6][7]. Хоча природний Cas9 вимагає складання керуючої РНК з двох докорінно різних РНК — CRISPR-РНК (crRNA), і транс-активаційну РНК (tracrRNA)[3], націлювання Cas9 було спрощено за допомогою вироблення єдиної химерної керуючої РНК. Передбачається, що Cas9 можна буде використовувати для зміни геному цілих популяцій організмів[8]. У 2015 році за допомогою Cas9 вперше був модифікований геном людського ембріона[9]. Розроблено технологію іммуногеномной інженерії гібридом, що дозволяє швидко перепрограмувати специфічність їх антитіл за допомогою Cas9[10].
MAGESTIC
Створена технологія, яка дозволяє редагувати окремі «літери» ДНК і РНК, що дозволить лікувати вроджені захворювання, викликані точковими мутаціями.[11] Створена технологія MAGESTIC (multiplexed accurate genome editing with short, trackable, integrated cellular barcodes), яка не тільки розщеплює ДНК, але ще і доставляє до місця розриву шматок ДНК необхідний для точної заміни (за допомогою гібридного зв'язанного ДНК білка LexA-Fkh1p), що підвищує точність і ефективність редагування[12][13].
Застосування dCas9 в епігенетиці
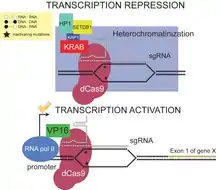
Поєднавши інактивовану молекулу dCas9, яка зв'язує ДНК але не розрізає її, з нуклеазами FokI, вдається отримати нуклеази і рестриктази для високоспецефічного розрізання ДНК[14][15][16]. Розроблено також спосіб виборчого епігенетичного перепрограмування активності генів за допомогою інактивованої молекули dCas9, з'єднаної з ферментом, що здійснює деметилювання ДНК[17]. Причому таке перепрограмування епігеному можна проводити навіть in vivo[18]. Пізніше з'ясувалося, що якщо вкоротити керуючу РНК до 14-15 нуклеотидів, то молекула Cas9 втрачає здатність розрізати ДНК[19][20]. Використовуючи цю властивість вдалося створити систему для виборчої активації певних генів in vivo і перевірити її ефективність шляхом лікування мишей зі змодельованими захворюваннями. У цього методу є тільки одна проблема: зазвичай система CRISPR завантажується в нешкідливий вірус, який називають аденоасоціованим вірусом (AAV), який переносить систему в клітину. Але весь білок, що складається з dCas9 і керуючої РНК, занадто великий, щоб поміститися в один AAV. Щоб вирішити цю проблему, дослідники завантажили dCas9 в один вірус, а керуючу РНК — в інший.
Нові способи доставки Cas9 в клітину
Основними вимогами до системи доставки Cas9 крім високої ефективності доставки є:
- конструкція, що синтезує Cas9 не повинна вбудовуватися в геном клітини і не повинна бути в клітині постійно щоб не заважати її роботі і не спровокувати імунних реакцій;
- спосіб доставки повинен бути здатний вмістити досить великий за розмірами фермент Cas9 або його кодуючу мРНК, а також одну або кілька керуючих РНК;
- вони повинні бути зручні для використання у вигляді ін'єкцій;
- такий спосіб разом з Cas9 і керуючими РНК має бути досить легко відтворюваним для великомасштабного виробництва лікарських препаратів для боротьби з поширеними хворобами.
Таким критеріям на відміну від вірусних систем доставки, відповідають ліпідні наночастинки. Так, наприклад, була створена біодеградуюча система доставки Cas9 ліпідною наночасткою, яка дозволила після одноразового введення досягти рівня понад 97 % інгібування одного з білків сироватки крові in vivo. Таке одноразове введення, незважаючи на тимчасовий характер системи доставки і компонентів системи редагування, призводило до тривалого пригнічення, що продовжувалося протягом 12 місяців[21].
Див. також
Примітки
- Human PubMed Reference:.
- Heler, Robert; Samai, Poulami; Modell, Joshua W.; Weiner, Catherine; Goldberg, Gregory W.; Bikard, David; Marraffini, Luciano A. (12 березня 2015). Cas9 specifies functional viral targets during CRISPR-Cas adaptation. Nature 519 (7542). с. 199–202. ISSN 1476-4687. PMC PMC4385744. PMID 25707807. doi:10.1038/nature14245. Процитовано 10 січня 2019.
- Charpentier, Emmanuelle; Doudna, Jennifer A.; Hauer, Michael; Fonfara, Ines; Chylinski, Krzysztof; Jinek, Martin (17 серпня 2012). A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science (англ.) 337 (6096). с. 816–821. ISSN 1095-9203. PMID 22745249. doi:10.1126/science.1225829. Процитовано 10 січня 2019.
- Komor, Alexis C.; Kim, Yongjoo B.; Packer, Michael S.; Zuris, John A.; Liu, David R. (05 19, 2016). Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533 (7603). с. 420–424. ISSN 1476-4687. PMC PMC4873371. PMID 27096365. doi:10.1038/nature17946. Процитовано 10 січня 2019.
- Church, George M.; Esvelt, Kevin M.; Mali, Prashant (2013-10). Cas9 as a versatile tool for engineering biology. Nature Methods (англ.) 10 (10). с. 957–963. ISSN 1548-7105. doi:10.1038/nmeth.2649. Процитовано 10 січня 2019.
- Church, George M.; Yang, Luhan; Kosuri, Sriram; Moosburner, Mark; Esvelt, Kevin M.; Stranges, P. Benjamin; Aach, John; Mali, Prashant (2013-09). CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nature Biotechnology (англ.) 31 (9). с. 833–838. ISSN 1546-1696. doi:10.1038/nbt.2675. Процитовано 10 січня 2019.
- Results for 'n2:1087-0156' > 'Journal, magazine' [WorldCat.org]. www.worldcat.org (англ.). Процитовано 10 січня 2019.
- Esvelt, Kevin M.; Smidler, Andrea L.; Catteruccia, Flaminia; Church, George M. (17 липня 2014). Emerging Technology: Concerning RNA-guided gene drives for the alteration of wild populations. eLife (англ.). Процитовано 10 січня 2019.
- Cyranoski, David; Reardon, Sara. Chinese scientists genetically modify human embryos. Nature News (англ.). doi:10.1038/nature.2015.17378. Процитовано 10 січня 2019.
- Reddy, Sai T.; Heuberger, Paul; Kelton, William J.; Parola, Cristina; Pogson, Mark (17 серпня 2016). Immunogenomic engineering of a plug-and-(dis)play hybridoma platform. Nature Communications (англ.) 7. с. 12535. ISSN 2041-1723. doi:10.1038/ncomms12535. Процитовано 10 січня 2019.
- Steinmetz, Lars M.; St.Onge, Robert P.; Salit, Marc L.; Haber, James E.; Bankaitis, Vytas A.; Davis, Ronald W.; Aiyar, Raeka S.; Wei, Wu та ін. (2018-06). Multiplexed precision genome editing with trackable genomic barcodes in yeast. Nature Biotechnology (англ.) 36 (6). с. 512–520. ISSN 1546-1696. doi:10.1038/nbt.4137. Процитовано 10 січня 2019.
- Разработана революционная технология точечного редактирования генома. www.infox.ru (рос.). Процитовано 10 січня 2019.
- Taking CRISPR from clipping scissors to word processor: New platform transforms gene editor into precision tool. ScienceDaily (англ.). Процитовано 10 січня 2019.
- Tsai, Shengdar Q.; Wyvekens, Nicolas; Khayter, Cyd; Foden, Jennifer A.; Thapar, Vishal; Reyon, Deepak; Goodwin, Mathew J.; Aryee, Martin J. та ін. (2014-6). Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing. Nature Biotechnology 32 (6). с. 569–576. ISSN 1546-1696. PMC PMC4090141. PMID 24770325. doi:10.1038/nbt.2908. Процитовано 10 січня 2019.
- Guilinger, John P.; Thompson, David B.; Liu, David R. (2014-6). Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nature Biotechnology 32 (6). с. 577–582. ISSN 1546-1696. PMC PMC4263420. PMID 24770324. doi:10.1038/nbt.2909. Процитовано 10 січня 2019.
- Wyvekens, Nicolas; Topkar, Ved V.; Khayter, Cyd; Joung, J. Keith; Tsai, Shengdar Q. (2015-7). Dimeric CRISPR RNA-Guided FokI-dCas9 Nucleases Directed by Truncated gRNAs for Highly Specific Genome Editing. Human Gene Therapy 26 (7). с. 425–431. ISSN 1557-7422. PMC PMC4509490. PMID 26068112. doi:10.1089/hum.2015.084. Процитовано 10 січня 2019.
- Xu, Xingxing; Tao, Yonghui; Gao, Xiaobo; Zhang, Lei; Li, Xufang; Zou, Weiguo; Ruan, Kangcheng; Wang, Feng та ін. (2016). A CRISPR-based approach for targeted DNA demethylation. Cell Discovery 2. с. 16009. ISSN 2056-5968. PMC PMC4853773. PMID 27462456. doi:10.1038/celldisc.2016.9. Процитовано 10 січня 2019.
- Morita, Sumiyo; Noguchi, Hirofumi; Horii, Takuro; Nakabayashi, Kazuhiko; Kimura, Mika; Okamura, Kohji; Sakai, Atsuhiko; Nakashima, Hideyuki та ін. (2016-10). Targeted DNA demethylation in vivo using dCas9-peptide repeat and scFv-TET1 catalytic domain fusions. Nature Biotechnology 34 (10). с. 1060–1065. ISSN 1546-1696. PMID 27571369. doi:10.1038/nbt.3658. Процитовано 10 січня 2019.
- Konermann, Silvana; Zhang, Feng; Gootenberg, Jonathan S.; Joung, Julia; Abudayyeh, Omar O.; Dahlman, James E. (2015-11). Orthogonal gene knockout and activation with a catalytically active Cas9 nuclease. Nature Biotechnology (англ.) 33 (11). с. 1159–1161. ISSN 1546-1696. doi:10.1038/nbt.3390. Процитовано 10 січня 2019.
- Church, George; Weiss, Ron; Collins, James J.; Ebrahimkhani, Mohammad R.; Kowal, Emma J. K.; Buchthal, Joanna; Vora, Suhani; Beal, Jacob та ін. (2015-11). Cas9 gRNA engineering for genome editing, activation and repression. Nature Methods (англ.) 12 (11). с. 1051–1054. ISSN 1548-7105. doi:10.1038/nmeth.3580. Процитовано 10 січня 2019.
- Morrissey, David V.; Chang, Yong; Strapps, Walter R.; Dombrowski, Christian; Harrington, William F.; Salomon, William E.; Wood, Kristy M.; Rohde, Ellen та ін. (27 лютого 2018). A Single Administration of CRISPR/Cas9 Lipid Nanoparticles Achieves Robust and Persistent In Vivo Genome Editing. Cell Reports (English) 22 (9). с. 2227–2235. ISSN 2211-1247. PMID 29490262. doi:10.1016/j.celrep.2018.02.014. Процитовано 10 січня 2019.