ДНК-полімераза II
ДНК-полімераза II (англ. DNA polymerase II, DNA Pol II або Pol II) - це прокаріотична ДНК-залежна ДНК-полімераза, що кодується геном PolB.[1]
.png.webp)
ДНК-полімераза II - це білок із молекулярною масою у 89,9 кДа, що відноситься до родини В ДНК-полімераз. Вперше фермент був виділений Томасом Корнбергом у 1970 році, та описаний ним же протягом наступних кількох років. [2][3][4] Питання функціональної активності полімерази в умовах in vivo досі обговорюється, однак вважається, що ДНК-полімераза II відіграє роль резервного ферменту у процесі прокаріотичної реплікації ДНК. Фермент має як здатність до 5′→3′ елонгації, так і 3′→5′ екзонуклеазну активність.[1]
Відкриття
ДНК-полімераза I була першою ДНК-залежною ДНК-полімеразою, виділеною з E.coli.[5] Кілька досліджень, що були проведені із використанням цього виділеного ферменту, показали, що ДНК-полімераза I бере участь у процесії репарації, і не є основною реплікативною полімеразою.[6] Щоб краще зрозуміти роль ДНК-полімерази I in vivo, у 1969 році вченими Де Люсія та Кернстом було створено мутанти E. coli з нестачею цього ферменту. [7] Цей мутантний штам був більш чутливим до дії ультрафіолетового світла. Саме це підтвердило гіпотезу, що роль ДНК-полімерази I полягає саме у репарації, а не реплікації. Мутантний штам мав здатність до росту з тією ж швидкістю, що й штами дикого типу, що вказувало на наявність іншого ферменту, відповідального за реплікацію ДНК. Виділення та опис цієї нової полімерази, що бере участь у напів консервативній реплікації ДНК, були проведені кількома лабораторіями паралельно.[2][4][3] Новий фермент був названий ДНК-полімеразою II, і деякий час вважалося, що він є основним ферментом реплікації E.coli.[8]
Структура
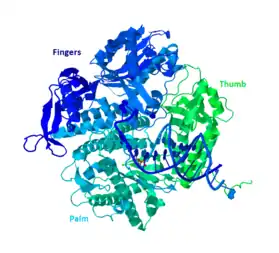
ДНК-полімераза II - це білок із молекулярною масою у 89,9 кДа, що складається із 783 амінокислот, і кодується геном PolB (dinA). ДНК-полімераза II є глобулярним білком, що функціонує як мономер, тоді як інші полімерази утворюють комплекси. Існує три основні частини цього мономеру, які разом інколи називають "рукою": долоня (palm), пальці (fingers) та великий палець (thumb). Полімеразний активний центр розташований у межах долоні - щілини, яка оточена двома характерними структурними доменами: рухливими пальцями (fingers) і великим пальцем (thumb). Великий палець взаємодіє з маленьким жолобком подвійної спіралі, утвореної матричним ланцюгом і таким, що синтезується. Пальці взаємодіють із матричним ланцюгом, який при цьому трохи вигинається навкруг пальців, експонуючи чергову азотисту основу для взаємодії з NTP. Сайт зв'язування NTP утворюється пальцями, залишками активного центру, нуклеотидом матричного ланцюга та 3'-кінцем ланцюга, що синтезується.[9] На долоні комплексу є три каталітичні центри, які будуть координуватися з двома іонами двовалентного металу, щоб функціонувати. ДНК-полімераза II має велику кількість копій у клітині - близько 30-50, тоді як рівень ДНК-полімерази III в клітині в п’ять разів менший.
Функції
Всі полімерази певним чином беруть участь у реплікації ДНК, синтезуючи ланцюги нуклеїнових кислот. Реплікація ДНК є важливим аспектом проліферації клітини. Без процесу реплікації клітина не може ділитися і передавати свою генетичну інформацію наступним поколінням. У прокаріотів, таких як E.coli, ДНК-полімераза III є основною полімеразою, яка бере участь у реплікації ДНК. Хоча ДНК-полімераза II не є основним фактором реплікації хромосом, вона виконує інші функції.
ДНК-полімераза II бере участь у реплікації ДНК. Не зважаючи на те, що ця полімераза не є такою швидкою, ДНК-полімераза III, вона має певні особливості. Цей фермент має асоційовану 3′→5′ екзонуклеазну активність поряд з активністю праймази. ДНК-полімераза II є високоточним ферментом з частотою помилок заміни ≤ 2×10 -6 і частотою помилок зсуву рамки зчитування ≤ 1×10 -6. ДНК-полімераза II може виправляти невідповідності, спричинені неточною роботою полімерази III. Також було показано, що ДНК-полімераза II бере участь у реплікації, однак вона залежить від типу ланцюга і переважно реплікує ланцюг, що відстає. На думку вчених, коли ДНК-полімераза III стає нефункціональною, тоді ДНК-полімераза II може бути залучена до точки реплікації і продовжує процес.[1]
Існує багато різних способів пошкодження ДНК, включаючи дію ультрафіолету та окислення, тому цілком логічно, що існують різні типи полімераз для усунення цих пошкоджень. Однією з важливих ролей ДНК-полімерази II є її здатність до відновлення міжланцюгових перехресних зшивок. Міжланцюгові перехресні зшивки виникають внаслідок дії на ДНК різних хімічних речовин, наприклад азотного іприту і псоралену. Усунути ці пошкодження важко, оскільки обидва ланцюги ДНК пошкоджені хімічним чинником, і генетична інформація на обох ланцюгах є ураженою. Точний механізм виправлення цих міжланцюгових перехресних зшивок все ще досліджується, але відомо, що полімераза II є основним ферментом у цьому процесі. [10]
ДНК-полімераза II досі не вивчена добре, тому існує багато можливих функцій цього ферменту, які потребують підтвердження [1]. Зокрема:
- відновлення ДНК після дії ультрафіолетового світла
- перезапуск процесу реплікації у E.coli після дії ультрафіолетового світла
- адаптивний мутагенез
- довгострокове виживання
Механізм дії
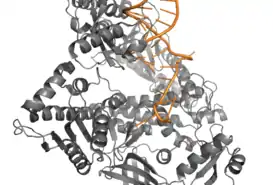
Під час реплікації ДНК основи можуть пошкоджуватись, що може призвести до зупинки реплікації. Щоб виправити помилку в послідовності, ДНК-полімераза II каталізує відновлення нуклеотидних пар основ. Дослідження in vitro показали, що полімераза II інколи взаємодіє з полімеразою III, щоб отримати доступ до ланцюга, що синтезується.[1][11][12] ДНК-полімераза II є також активною під час реплікації ДНК, оскільки будь-які помилки, що виникають внаслідок роботи полімерази III, будуть у ланцюзі, що синтезується, а не в консервативному. N-кінцевий домен ДНК-полімерази II відповідає за асоціацію та дисоціацію ланцюга ДНК з каталітичною субодиницею. Швидше за все, в N-кінцевому домені ДНК-полімерази II є дві ділянки, які розпізнають одноланцюгову ДНК. Один сайт відповідає за приєднання одноланцюгової ДНК до ДНК-полімерази II, а інший сайт - за від'єднання. [13]
.png.webp)
Після приєднання субстрату, ДНК-полімераза II зв’язує NTP для підтримки водневих зв'язків у структурі ДНК. Потім зв’язується правильний, непошкоджений нуклеотид (dNTP), і ферментний комплекс зазнає конформаційних змін субдоменів та амінокислотних залишків. Ці конформаційні зміни дозволяють прискорити процес репарації.[14] Активний центр ферменту містить два іони Mg2+ , які стабілізуються каталітичними аспарагіновими кислотами D419 і D547.[15] Іони магнію зв’язуються з ДНК разом з dNTP у відкритому стані та координують конформаційні зміни амінокислотних залишків активного центру для того, щоб відбувався каталіз (закритий стан). Після вивільнення іонів магнію фермент повертається у відкритий стан.[16]
Регуляція
Кількість ДНК-полімерази II в клітині зазвичай у п’ять разів перевищує кількість полімерази III. Це дозволяє полімеразі II за своїми функціями випереджувати дію полімерази III у разі помилкового утворення пар основ. Кількість полімерази II може зростати при індукції SOS-реакції, яка активізує ген polB, тому кількість ферменту збільшується приблизно в сім разів. Хоча полімераза II може працювати на обох ланцюгах, було показано, що вона більше виконує свою роль на ланцюзі, що відстає. [1]
Посилання
- Banach-Orlowska, Magdalena; Fijalkowska, Iwona J.; Schaaper, Roel M.; Jonczyk, Piotr (2005). DNA polymerase II as a fidelity factor in chromosomal DNA synthesis in Escherichia coli. Molecular Microbiology (англ.) 58 (1). с. 61–70. ISSN 1365-2958. doi:10.1111/j.1365-2958.2005.04805.x. Процитовано 1 грудня 2021.
- Kornberg, Thomas; Gefter, Malcolm L. (30 вересня 1970). DNA synthesis in cell-free extracts of a DNA polymerase-defective mutant. Biochemical and Biophysical Research Communications (англ.) 40 (6). с. 1348–1355. ISSN 0006-291X. doi:10.1016/0006-291X(70)90014-8. Процитовано 1 грудня 2021.
- Moses, Robb E.; Richardson, Charles C. (24 грудня 1970). A new DNA polymerase activity of Escherichia coli. I. Purification and properties of the activity present in E. coli polAl. Biochemical and Biophysical Research Communications (англ.) 41 (6). с. 1557–1564. ISSN 0006-291X. doi:10.1016/0006-291X(70)90565-6. Процитовано 1 грудня 2021.
- Knippers, Rolf (1970-12). DNA Polymerase II. Nature (англ.) 228 (5276). с. 1050–1053. ISSN 1476-4687. doi:10.1038/2281050a0. Процитовано 1 грудня 2021.
- Lehman, I.R.; Bessman, Maurice J.; Simms, Ernest S.; Kornberg, Arthur (1958-07). Enzymatic Synthesis of Deoxyribonucleic Acid. Journal of Biological Chemistry 233 (1). с. 163–170. ISSN 0021-9258. doi:10.1016/s0021-9258(19)68048-8. Процитовано 1 грудня 2021.
- Smith, Douglas W.; Schaller, Heinz E.; Bonhoeffer, Friedrich J. (1970-05). DNA Synthesis in vitro. Nature (англ.) 226 (5247). с. 711–713. ISSN 1476-4687. doi:10.1038/226711a0. Процитовано 1 грудня 2021.
- De Lucia, Paula; Cairns, John (1969-12). Isolation of an E. coli Strain with a Mutation affecting DNA Polymerase. Nature (англ.) 224 (5225). с. 1164–1166. ISSN 1476-4687. doi:10.1038/2241164a0. Процитовано 1 грудня 2021.
- Kornberg, Thomas; Gefter, Malcolm L. (1 квітня 1971). Purification and DNA Synthesis in Cell-Free Extracts: Properties of DNA Polymerase II. Proceedings of the National Academy of Sciences (англ.) 68 (4). с. 761–764. ISSN 0027-8424. PMC PMC389037. PMID 4927672. doi:10.1073/pnas.68.4.761. Процитовано 1 грудня 2021.
- Сиволоб, А.В. (2008). Молекулярна біологія (українська). Київ: Видавничо-поліграфічний центр "Київський університет". с. 384. ISBN 978-966-439-068-9.
- Bebenek, Katarzyna; Kunkel, Thomas A. (1 січня 2004). Functions of DNA Polymerases. Advances in Protein Chemistry (англ.) 69. Academic Press. с. 137–165. doi:10.1016/s0065-3233(04)69005-x.
- Wickner, Sue; Hurwitz, Jerard (1 жовтня 1974). Conversion of ϕX174 Viral DNA to Double-Stranded Form by Purified Escherichia coli Proteins. Proceedings of the National Academy of Sciences (англ.) 71 (10). с. 4120–4124. ISSN 0027-8424. PMC PMC434340. PMID 4610569. doi:10.1073/pnas.71.10.4120. Процитовано 1 грудня 2021.
- Bonner, C. A.; Stukenberg, P. T.; Rajagopalan, M.; Eritja, R.; O'Donnell, M.; McEntee, K.; Echols, H.; Goodman, M. F. (5 червня 1992). Processive DNA synthesis by DNA polymerase II mediated by DNA polymerase III accessory proteins.. Journal of Biological Chemistry (English) 267 (16). с. 11431–11438. ISSN 0021-9258. PMID 1534562. doi:10.1016/S0021-9258(19)49928-6. Процитовано 1 грудня 2021.
- Maki, Satoko; Hashimoto, Keiji; Ohara, Takeshi; Sugino, Akio (14 серпня 1998). DNA Polymerase II (ε) of Saccharomyces cerevisiaeDissociates from the DNA Template by Sensing Single-stranded DNA *. Journal of Biological Chemistry (English) 273 (33). с. 21332–21341. ISSN 0021-9258. PMID 9694894. doi:10.1074/jbc.273.33.21332. Процитовано 1 грудня 2021.
- Beard, William A.; Wilson, Samuel H. (6 травня 2014). Structure and Mechanism of DNA Polymerase β. Biochemistry 53 (17). с. 2768–2780. ISSN 0006-2960. PMC PMC4018062. PMID 24717170. doi:10.1021/bi500139h. Процитовано 1 грудня 2021.
- Wang, Feng; Yang, Wei (24 грудня 2009). Structural Insight Into Translesion Synthesis By DNA Pol II. Cell 139 (7). с. 1279–1289. ISSN 0092-8674. PMC 3480344. PMID 20064374. doi:10.1016/j.cell.2009.11.043. Процитовано 1 грудня 2021.
- Yang, Linjing; Arora, Karunesh; Beard, William A.; Wilson, Samuel H.; Schlick, Tamar (14 липня 2004). Critical Role of Magnesium Ions in DNA Polymerase β's Closing and Active Site Assembly. Journal of the American Chemical Society 126 (27). с. 8441–8453. ISSN 0002-7863. doi:10.1021/ja049412o. Процитовано 1 грудня 2021.
