Deinococcus radiodurans
Deinococcus radiodurans — грам-позитивна, екстремофільна кокова бактерія роду Deinococcus, один з найстійкіших організмів до дії іонізуючого випромінювання.[1] Вперше був виділений з консервованого м'яса, підданого дії гамма-випромінювання з метою вивчення можливості стерилізації.[2] Організм був вперше описаний в 1960 році під назвою Micrococcus radiodurans[3], а в 1981 році переведений до новоствореного роду Deinococcus.[4] Зараз розроблюються способи використання D. radiodurans в біоочищенні радіоактивно заражених стічних вод.
? Deinococcus radiodurans | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
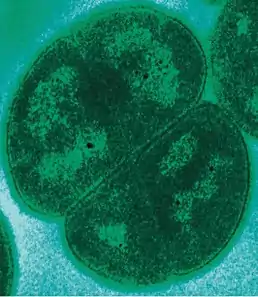 Тетрада D. radiodurans | ||||||||||||||
| Біологічна класифікація | ||||||||||||||
| ||||||||||||||
| Deinococcus radiodurans (ex Raj et al. 1960) Brooks et Murray 1981 | ||||||||||||||
Посилання | ||||||||||||||
| ||||||||||||||
Біологічні властивості
Морфологія
Забарвлюється за методу Грама позитивно, хоча клітинна стінка має будову, типову для грам-негативних бактерій[5][6]), це нерухомий кок діаметром 1,5—3,5 мікрон. На мікроскопічних препаратах розташовується по дві або частіше чотири клітки, утворюючи тетради. Не утворює капсул і спор. синтезує червоний пігмент.[7]
Культуральні властивості
D. radiodurans — хемоорганогетеротроф, облігатний аероб. Росте на простих живильних середовищах. На агаризованих живильних середовищах утворює гладкі, опуклі колонії від рожевого до червоного кольору[8]. Штами D. radiodurans виділялися з великої різноманітності субстратів: послід слонів, ґрунти до арктичних глибин і пісків пустель[9][10], тому не можна говорити про специфічне місце проживання цього мікроорганізму.[11]
Геном
Унікальною особливістю геному D. radiodurans є те, що кожна кільцева молекула ДНК геному представлена в кількох копіях, що разом утворюють переплетені кільця, кожне кільце містить по кілька копій однієї молекули ДНК.[12] Іншою унікальною особливістю D. radiodurans є наявність РНК-лігаз, здатних зшивати молекули РНК в гібридному комплексі РНК-ДНК[13][14]. Геном D. radiodurans штаму R1 представлений чотирма молекулами ДНК: дві хромосоми і дві плазміди — мегаплазміда і мала плазміда.[15] Хромосома 1 D. radiodurans є кільцевою дволанцюжковою молекулою ДНК розміром 2648638 пар основ та містить 2687 генів, з яких 2629 кодують білки.[16] Хромосома 2 менше розмірами (412348 пар основ) і містить 369 генів, з яких 368 кодують білки[17]. Мегаплазміда MP1 є дволанцюжковою кільцевою молекулою ДНК розміром 177466 пар основ і містить 148 генів, з яких 145 кодують білки.[18] Плазміда CP1 також представлена кільцевою молекулою ДНК, розміром 45704 пар основ, і містить 40 генів, з яких 39 кодують білки.[19] Відомо також кілька плазмід, що впливають на резистентність до лізоциму і допустиму температуру росту.[20] Мікроорганізм має природну компетентність до трансформації чужорідної ДНК[21]. Геном D. radiodurans вельми близький до геному Thermus aquaticus і порівняння геномів показує дивергентні шляхи до адаптації до термофільності і стійкості до радіації.[22][23]
Стійкість до дії іонізуючого випромінювання
D. radiodurans широко відома своєю високою стійкістю до дії радіації та є одним з найстійкіших до дії радіації організмом у світі — D. radiodurans здатний виживати при дозі до 10000 Гр. (для людини летальна доза радіації становить близько 5 Гр., для Escherichia coli — 2000 Гр.).[24]
Ймовірно, висока стійкість до дії іонізуючого випромінювання виникла внаслідок виникнення стійкості до висушування, оскільки механізми пошкодження ДНК, а отже і стійкості до радіації і висушування, східні[25], до того ж D. radiodurans синтезує так звані LEA-білки, що запобігають агрегації білків під час висушування[26]. Довгий час такий рівень стійкості до дії радіації був не зовсім зрозумілий. Зараз відомо, що D. radiodurans зберігає в клітині по кілька копій геному, упакованих у вигляді торів або кілець[27], додаткові копії геному дозволяють в точності відновити геном після численних одно- і дволанцюжкових розривів. Було також показано, що як мінімум дві копії геному при масованих дволанцюжкових розривах утворюють повний геном при реасоціації фрагментів ДНК, що утворилися, після чого проводить повторний синтез пошкоджених ділянок з гомологічних непошкоджених послідовностей, при цьому утворюється D-петля та відбувається рекомбінація між гомологічними послідовностями шляхом RecA-залежної гомологічної рекомбінації.[28]
RecA D. radiodurans може експресуватися тільки в клітинах свого виду, для E.coli, наприклад, він надає летальну дію[29]. Певну роль в резистентності до дії радіації надає також присутність особливого білка, що зв'язується з одноланцюжковою ДНК і ймовірно грає роль в реплікації пошкодженої ділянки.[30] На радіорезистентність впливає також синтез білка DdrA, що забезпечує цілісність геному.[31] Білок IrrE, регулятор експресії гену recA, також впливає на рівень стійкості до дії радіації.[32] Мікроорганізм має рибонуклеопротеїни, що також надають дію на стійкість бактерії до ультрафіолетового випромінювання.[33] Для захисту від окислювального стресу, що зазвичай супроводжує дію іонізуючого випромінювання, D. radiodurans використовує особливий фермент тіоредоксин-редуктазу[34], а також синтезує супероксиддісмутазу.[35]
Використання
Стійкість до дії радіації D. radiodurans унікальні, мікроорганізм також вельми стійкий до несприятливих умов навколишнього середовища, що робить його придатним для біоочищення радіоактивних відходів. Зараз ведуться дослідження із застосування D. radiodurans у біоочищенні радіоактивних забруднень, зокрема розчинених іонів ртуті.[36]
Посилання
- Rainey FA, Ray K, Ferreira M, Gatz BZ, Nobre MF, Bagaley D, Rash BA, Park MJ, Earl AM, Shank NC, Small AM, Henk MC, Battista JR, Kämpfer P, da Costa MS (2005). Extensive diversity of ionizing-radiation-resistant bacteria recovered from Sonoran Desert soil and description of nine new species of the genus Deinococcus obtained from a single soil sample. Appl Environ Microbiol. 71 (9): 5225–35. PMID 16151108.
- Anderson A W, Nordan H. C., Cain R. F., Parrish G., Duggan D. Studies on а radio-resistant micrococcus. I. Isolation, morphology, cultural characteristics, and resistance to gamma radiation// Food Technol. 1956 № 10 Р. 575—577
- Raj H.D., Duryee F.L., Deeney A.M., Wang C.H., Anderson A.W., Elliker P.R. Utilization of carbohydrates and amino acids by Micrococcus radiodurans.// Can. J. Microbiol., 1960 #6 P/ 289—298
- http://ijs.sgmjournals.org/cgi/content/abstract/31/3/353%5Bнедоступне+посилання+з+червня+2019%5D
- Embley, T. M., A. G. O'Donnell, R. Wait, and J. Rostron. Lipid and cell wall amino acid composition in the classification of members of the genus Deinococcus.// Syst. Appl. Microbiol. 1987. № 10 Р. 20-27
- http://www.ncbi.nlm.nih.gov/pubmed/5882498?dopt=Abstract
- http://www.ebi.ac.uk/2can/genomes/bacteria/Deinococcus_radiodurans.html
- <http://www.biology.lsu.edu/webfac/jbattista/publications/downloads/Against_All_Odds.pdf/ Архівовано 17 грудня 2008 у Wayback Machine.
- Ito H, Iizuka H, Takehisa M, Watanabe H. Isolation and identification of radiation-resistant cocci belonging to genus Deinococcus from sewage sludges and animal feeds// Agric Biol Chem., 1983 № 47 Р. 1239—1247
- http://www.ncbi.nlm.nih.gov/pubmed/5866032
- http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=91106#B1
- Архівована копія. Архів оригіналу за 3 лютого 2012. Процитовано 24 січня 2009.
- http://www.jbc.org/cgi/content/abstract/279/49/50654
- http://nar.oxfordjournals.org/cgi/content/abstract/35/3/839
- http://www.ncbi.nlm.nih.gov/pubmed/10567266?ordinalpos=1&itool=EntrezSystem2.PEntrez.Pubmed.Pubmed_ResultsPanel.Pubmed_RVDocSum
- http://www.ncbi.nlm.nih.gov/sites/entrez?db=genome&cmd=Retrieve&dopt=Overview&list_uids=144
- http://www.ncbi.nlm.nih.gov/sites/entrez?db=genome&cmd=Retrieve&dopt=Overview&list_uids=145
- http://www.ncbi.nlm.nih.gov/sites/entrez?db=genome&cmd=Retrieve&dopt=Overview&list_uids=15154
- http://www.ncbi.nlm.nih.gov/sites/entrez?db=genome&cmd=Retrieve&dopt=Overview&list_uids=15155
- http://www.jstor.org/pss/3578742
- http://www.pnas.org/content/61/1/176.full.pdf+html?ijkey=58033a47bbae803c7264d9b24e0541d24bef123b&keytype2=tf_ipsecsha
- http://www.biomedcentral.com/1471-2148/5/57
- http://mmbr.asm.org/cgi/content/full/65/1/44
- http://www.usuhs.mil/pat/deinococcus/index_20.htm
- http://jb.asm.org/cgi/reprint/178/3/633?view=long&pmid=8550493
- http://www.biochemj.org/bj/388/0151/bj3880151.htm
- http://www.sciencemag.org/cgi/content/abstract/299/5604/254
- http://www.insermactualites.net/fileadmin/user_upload/202/fichiers/nature05160Radman.pdf%5Bнедоступне+посилання+з+лютого+2019%5D
- http://jb.asm.org/cgi/content/abstract/178/1/130
- http://www.pnas.org/content/101/23/8575.abstract
- http://biology.plosjournals.org/perlserv/?request=get-document&doi=10.1371/journal.pbio.0020304&ct=1
- http://jb.asm.org/cgi/content/abstract/184/22/6216
- http://genesdev.cshlp.org/cgi/content/abstract/14/7/777
- http://www.ncbi.nlm.nih.gov/pubmed/16880549?dopt=Abstract
- http://www.ncbi.nlm.nih.gov/pubmed/15807266?dopt=Abstract
- http://www.ncbi.nlm.nih.gov/pubmed/10625398