МікроРНК
МікроРНК (англ. microRNA, miRNA) — клас некодуючих молекул РНК, довжиною ~22 нуклеотидів, що беруть участь у регуляції трансляції та деградації мРНК. Особливістю мікроРНК є неповна комплементарність їх взаємодії з цільовою мРНК.[1][2]. Станом на 2014 р. всього зареєстровано 28 645 пре-мікроРНК з різних тварин, рослин і вірусів (1881 людини) і 35 822 зрілих мікроРНК (2588 людини).[3][4] Довжина зареєстрованих зрілих молекул: 15–34 нуклеотидів.
Походження
МікроРНК кодуються генами, перші з яких були виявлені у 1993 році у Caenorhabditis elegans.[5] Еволюційно гени мікроРНК є похідними мобільних елементів класу MITE (англ. miniature inverted-repeat transposable elements) та виникли з окремих копій транспозонів. Транскрипти MITE завдяки наявності інвертованих повторів та формуванню шпильок клітина розпізнавала та використовувала для пригнічення активності решти копій транспозонів. На сьогодні мікроРНК виявлені у рослинних й у тваринних організмах. Мішенями мікроРНК є значна кількість генів — щонайменше третина генів генома. Раніше вважалось, що мікроРНК наявні лише у багатоклітинних організмах, але, наявність цієї групи молекул виявлена і у одноклітинних еукаріотів, а саме — у зелених водоростей Chlamydomonas reinhardtii. Це свідчить про великий еволюційний вік мікроРНК.
Будова і процесинг
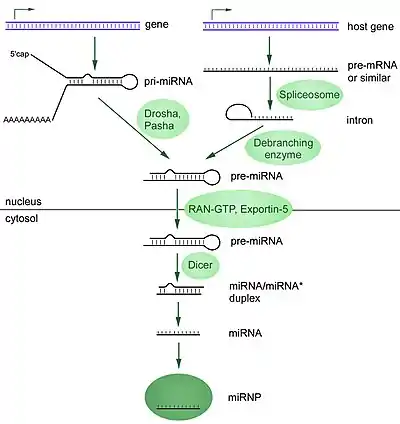
Гени мікроРНК транскрибуються із утворенням довгих транскриптів первинної мікроРНК (англ. primordial miRNA). Ці РНК процесуються у ядрі, внаслідок чого вони перетворюються у пре-мікроРНК — структури у вигляді шпильки довжиною приблизно 70 нуклеотидів. Комплекс процесингу первинного транскрипту в пре-мікроРНК містить фермент із активністю РНКази ІІІ, який називають Drosha, та білок, що зв'язує дволанцюгову РНК — Pasha. Цей комплекс відрізає довгі «хвости» молекули РНК, залишаючи невелику дволанцюгову молекулу зі шпилькою, довжиною 70-90 нуклеотидів — пре-мікроРНК. Після утворення пре-мікроРНК транспортується з ядра в цитоплазму, де від неї відрізується шпилька за допомогою ферменту дайсер (у Drosophila melanogaster та С.elegans для міРНК та мікроРНК існують різні ізоформи ферменту дайсер[6]). У багатьох, але не у всіх, випадках тільки один з ланцюгів зрілої мікроРНК залишається в цитоплазмі і зв'язується із ферментативним RISC-комплексом[7]. Другий (пасажирський) ланцюг деградується РНКазами. Відомий також шлях процесингу мікроРНК, незалежний від дайсеру. У цьому випадку пре-мікроРНК ріжеться білком аргонавт 2.[8]
Функція
Принципова різниця між міРНК та мікроРНК полягає у тому, що у тварин послідовність мікроРНК не повністю комплементарна до послідовності мРНК-мішені, таким чином мікроРНК можуть інгібувати трансляцію із кількох різних мРНК, що містять схожі послідовності (у рослин як мікроРНК так і міРНК зазвичай повністю комплементарні до РНК-мішені). МікроРНК приєднуються до 3'-UTR (3'-кінцевої ділянки, що не транслюється) мРНК і викликають дестабілізацію комплексу ініціації трансляції на 5'-UTR[9].
МікроРНК відіграють важливу роль у пригніченні експресії інших генів та у регуляції розвитку, особливо у визначенні часу морфогенезу та підтриманні недиференційованих або не повністю диференційованих типів клітин, таких як стовбурові клітини.
Позаклітинні мікроРНК
Зрілі мікроРНК існують не тільки в цитоплазмі, а також і в біологічних рідинах: плазмі крові, слині, сльозах, спинномозковій рідині, сечі, грудному молоці, молозиві, амніотичній рідині, семенній рідині, рідині порожнини живота, плевральній рідині, а також в калі.[10][11] Позаклітинні мікроРНК дуже стабільні тому що знаходяться в екзосомах, або зв'язані з білком аргонавт або ліпопротеїнами високої щільності. Починаючи з 2008 р. багато зусиль було покладено на пошук кореляції між спектром циркуляційних мікроРНК і раковими чи іншими захворюваннями, але встановити надійних біомаркерів поки не вдалося.[12][13][14] Не знайшла достатньої підтримки екзосомна модель міжклітинних комунікацій за рахунок мікроРНК.[15][16] Порівняння спектрів мікроРНК в грудному молоці людей та інших ссавців виявило спільні типи зустрінутих мікроРНК, але можливий вплив молочної мікроРНК на розвиток новонароджених ще не знайдено.[17]
МікроРНК і мікробіота
У 2016 р. гарвардські вчені відкрили механізм формування флори кишечника через потрапляння фекальних мікроРНК, які виділяються епітелієм, всередину бактерій і регуляцію їх генів.[18] Виявилось, що специфічний набір мікроРНК виробляється та експортується в порожнину кишечника клітинами з поверхні епітелію, а також стовбуровими клітинами, які знаходяться в +4 шарі, разом з їх більш диференційованими нащадками з характерною експресією гена Hopx. Концентрація мікроРНК в клубовій кишці більша ніж у товстій. Спектр типів мікроРНК в різних секціях травного тракту змінюється. Тести з найпоширенішими типами мікроРНК показали, що hsa-miR-515-5p прискорює ріст кишечної бактерії Fusobacterium nucleatum специфічно діючи на її 16S рибосомну РНК. Інша мікроРНК — hsa-miR-1226-5p прискорює ріст E. coli через взаємодію з мРНК транспортного білка YegH. Лабораторні миші з порушенням у виробництві фекальних мікроРНК дуже вразливі до коліту, але якщо їх годувати цими мікроРНК, то вдається зменшити наслідки патології кишечника. Наступним кроком буде розробка лікувальних засобів на основі мікроРНК.[19][20]
Примітки
- Wang QL, Li ZH (2007). The functions of microRNAs in plants. Front. Biosci. 12: 3975–82. PMID 17485351.
- Zhao Y, Srivastava D (2007). A developmental view of microRNA function. Trends Biochem. Sci. 32 (4): 189–97. PMID 17350266. doi:10.1016/j.tibs.2007.02.006.
- Kozomara A, Griffiths-Jones S (2014). miRBase: annotating high confidence microRNAs using deep sequencing data. Nucl Acids Res 42 (D1): D68–D73. PMID 24275495. doi:10.1093/nar/gkt1181.
- miRBase: the microRNA database. Манчестерський університет. Процитовано 17 січня 2016.
- The microRNAs of Caenorhabditis elegans http://cseweb.ucsd.edu/classes/wi05/cse206b/pub/burge_MiRscan.pdf
- Lee Y, Nakahara K, Pham J, Kim K, He Z, Sontheimer E, Carthew R (2004). Distinct roles for Drosophila Dicer-1 and Dicer-2 in the siRNA/miRNA silencing pathways. Cell 117 (1): 69–81. PMID 15066283. doi:10.1016/S0092-8674(04)00261-2.
- Gregory R, Chendrimada T, Shiekhattar R (2006). MicroRNA biogenesis: isolation and characterization of the microprocessor complex. Methods Mol Biol 342: 33–47. PMID 16957365. doi:10.1385/1-59745-123-1:33.
- Cheloufi S, Dos Santos CO, Chong MM, Hannon GJ. A dicer-independent miRNA biogenesis pathway that requires Ago catalysis // Nature. — 2010. — Вип. doi:10.1038/nature09092. — С. Published Online April 27, 2010.
- Pillai RS, Bhattacharyya SN, Filipowicz W (2007). Repression of protein synthesis by miRNAs: how many mechanisms?. Trends Cell Biol 17 (3): 118–26. PMID 17197185. doi:10.1016/j.tcb.2006.12.007.
- Weber JA, Baxter DH, Zhang S, Huang DY, Huang KH, Lee MJ, Galas DJ, Wang K (2010). The microRNA spectrum in 12 body fluids. Clin Chem 56 (11): 1733–1741. PMID 20847327. doi:10.1373/clinchem.2010.147405.
- Ahmed FE, Jeffries CD, Vos PW, Flake G, Nuovo GJ, Sinar DR, Naziri W, Marcuard SP (2009). Diagnostic microRNA markers for screening sporadic human colon cancer and active ulcerative colitis in stool and tissue. Cancer Genomics Proteomics 6 (5): 281–295. PMID 19996134.
- Lawrie CH, Gal S, Dunlop HM, Pushkaran B, Liggins AP, Pulford K, Banham AH, Pezzella F, Boultwood J, Wainscoat JS, Hatton CS, Harris AL (2008). Detection of elevated levels of tumour-associated microRNAs in serum of patients with diffuse large B-cell lymphoma. Br J Haematol 141 (5): 672–675. PMID 18318758. doi:10.1111/j.1365-2141.2008.07077.x.
- Leidner RS, Li L, Thompson CL (2013). Dampening Enthusiasm for Circulating MicroRNA in Breast Cancer. PLoS One 8 (3): e57841. PMID 23472110. doi:10.1371/journal.pone.0057841.
- Angelini TG, Emanueli C (2015). MicroRNAs as clinical biomarkers?. Front Genet 6: 240. PMID 26236333. doi:10.3389/fgene.2015.00240.
- Chevillet JR, Kang Q, Ruf IK, Briggs HA, Vojtech LN, Hughes SM, Cheng HH, Arroyo JD, Meredith EK, Gallichotte EN, Pogosova-Agadjanyan EL, Morrissey C, Stirewalt DL, Hladik F, Yu EY, Higano CS, Tewari M (2014). Quantitative and stoichiometric analysis of the microRNA content of exosomes. Proc Natl Acad Sci USA 111 (41): 14888–14893. PMID 25267620. doi:10.1073/pnas.1408301111.
- Turchinovich A1, Tonevitsky AG, Cho WC, Burwinkel B (2015). Check and mate to exosomal extracellular miRNA: new lesson from a new approach. Front Mol Biosci 2: 11. PMID 25988178. doi:10.3389/fmolb.2015.00011.
- Simpson MR, Brede G, Johansen J, Johnsen R, Storrø O, Sætrom P, Øien T (2015). Human Breast Milk miRNA, Maternal Probiotic Supplementation and Atopic Dermatitis in Offspring. PLoS One 10 (12): e0143496. PMID 26657066. doi:10.1371/journal.pone.0143496.
- Liu S, da Cunha AP, Rezende RM, Cialic R, Wei Z, Bry L, Comstock LE, Gandhi R, Weiner HL (2016). The Host Shapes the Gut Microbiota via Fecal MicroRNA. Cell Host Microbe 19 (1): 32–43. PMID 26764595. doi:10.1016/j.chom.2015.12.005.
- Maron DF (2016). Does our Microbiome Control Us or Do We Control It?. Scientific American.
- Mammals shape their microbiome to prevent disease (прес-реліз). Cell Press. 13 січня 2016. Процитовано 17 січня 2016.
