Хімічна біологія
Хімічна біологія — це наукова дисципліна, що охоплює галузі хімії та біології . Дисципліна передбачає застосування хімічних методик, аналізу, а часто і малих молекул, отриманих за допомогою синтетичної хімії, для вивчення та маніпулювання біологічними системами. На відміну від біохімії, яка передбачає вивчення хімії біомолекул та регулювання біохімічних процесів всередині клітин і між ними, хімічна біологія займається хімією, застосованою до біології (синтез біомолекул, моделювання біологічних систем тощо).
Вступ
Деякі форми хімічної біології намагаються відповісти на біологічні питання, безпосередньо досліджуючи живі системи на хімічному рівні. На відміну від досліджень, що використовують біохімію, генетику чи молекулярну біологію, де мутагенез може забезпечити нову цікаву для організму, клітину чи біомолекул цікаву систему хімічної біології зондує in vitro та in vivo з невеликими молекулами, розробленими для конкретних призначення або визначені на основі біохімічного або клітинного скринінгу (див. хімічну генетику).
Хімічна біологія є однією з декількох міждисциплінарних наук, які мають тенденцію відрізнятися від старих, редукціоністських галузей і цілями яких є досягнення опису наукового цілісності. Хімічна біологія має наукові, історичні та філософські корені в медичній хімії, надмолекулярній хімії, біоорганічній хімії, фармакології, генетиці, біохімії та метаболічній інженерії.
Системи, що цікавлять
Методи збагачення протеоміки
Хімічні біологи працюють над покращенням протеоміки шляхом розробки стратегій збагачення, хімічних позначок спорідненості та нових зондів. Зразки для протеоміки часто містять безліч пептидних послідовностей, і послідовність, що представляє інтерес, може бути сильно представлена або має малу кількість, що створює бар'єр для їх виявлення. Хімічні біологічні методи можуть зменшити складність зразка шляхом селективного збагачення за допомогою афінної хроматографії . Це включає націлювання на пептид із відмітною ознакою, такою як етикетка біотіну або пост-трансляційна модифікація .[1] Розроблені методи, які включають використання антитіл, лектинів для захоплення глікопротеїнів, а також іммобілізованих іонів металів для захоплення фосфорильованих пептидів та ферментних субстратів для захоплення виділених ферментів.
Ферментні зонди
Для дослідження ферментативної активності на відміну від загального білка були розроблені реагенти на основі активності для маркування ферментативно активної форми білків (див. Протеоміка). Наприклад, інгібітори протеїнози серин-гідролази та цистеїну були перетворені на інгібітори самогубств. [2] Ця стратегія розширює можливість вибіркового аналізу складових частин малого ряду шляхом прямого націлювання. [3] Активність ферменту також може контролюватися через перетворений субстрат. [4] Ідентифікація ферментних субстратів є проблемою значних труднощів протеоміки і є життєво важливою для розуміння шляхів передачі сигналу в клітинах. Розроблений метод використовує «аналого-чутливі» кінази для маркування субстратів, використовуючи неприродний аналог АТФ, полегшуючи візуалізацію та ідентифікацію за допомогою унікальної ручки. [5]
Глікобіологія
Хоча ДНК, РНК і білки кодуються на генетичному рівні, глікани (полімери цукру) не кодуються безпосередньо з геному, і для їх вивчення доступно менше інструментів. Отже, глікобіологія — область активних досліджень для хіміків-біологів. Наприклад, клітини можуть забезпечуватися синтетичними варіантами природних цукрів для перевірки їх функції. Дослідницька група Кероліна Бертоцці розробила методи для реакції молекул на поверхні клітин за допомогою синтетичних цукрів.[2]
Комбінаторна хімія
Хімічні біологи використовували автоматизований синтез різних бібліотек з невеликими молекулами для того, щоб провести високопропускний аналіз біологічних процесів. Такі експерименти можуть призвести до виявлення невеликих молекул з антибіотичними чи хіміотерапевтичними властивостями. Ці комбінаторні підходи з хімії ідентичні тим, які застосовуються в галузі фармакології.
Використовуючи біологію
Багато дослідницьких програм також зосереджені на використанні природних біомолекул для виконання біологічних завдань або підтримки нового хімічного методу. У зв'язку з цим дослідники хімічної біології показали, що ДНК може слугувати шаблоном для синтетичної хімії, самозбірні білки можуть служити структурним каркасом нових матеріалів, а РНК може розвиватися in vitro для отримання нової каталітичної функції. Крім того, гетеробіфункціональні (двосторонні) синтетичні малі молекули, такі як димеризатори або PROTAC, об'єднують два білки всередині клітин, що може синтетично індукувати важливі нові біологічні функції, такі як цільова деградація білка.[3]
Синтез пептидів
Хімічний синтез білків є цінним інструментом у хімічній біології, оскільки він дозволяє вводити неприродні амінокислоти, а також залишкові специфічні включення " посттрансляційних модифікацій ", таких як фосфорилювання, глікозилювання, ацетилювання та навіть повсюднення . Ці можливості цінні для хіміків-біологів, оскільки неприродні амінокислоти можуть використовуватися для зондування та зміни функціональності білків, тоді як після трансляційні модифікації широко відомі для регулювання структури та активності білків. Хоча для досягнення цих цілей були розроблені суто біологічні методи, хімічний синтез пептидів часто має менший технічний та практичний бар'єр для отримання невеликих кількостей бажаного білка.
Для того, щоб зробити поліпептидні ланцюги розміром з білками через дрібні пептидні фрагменти, зроблені синтезом, хімічні біологи використовують процес нативної хімічної перев'язки .[4] Нативні хімічні перев'язки включають в себе зв'язування С-кінцевого тиоестеру і N-кінцевого цистеїнового залишку, що в кінцевому рахунку призводить до утворення «нативної» амідної зв'язку. Інші стратегії, які використовувались для лігування пептидних фрагментів за допомогою хімії перенесення ацилу, вперше введеної з нативною хімічною лігацією, включають експресовану лігування білка,[5] методи сульфурації / десульфурації[6] та використання знімних допоміжних речовин тиолу.[7] Лігація експресованого білка дозволяє здійснити біотехнологічну установку С-кінцевого тиоестеру з використанням інтейн, тим самим дозволяючи приєднання синтетичного N-кінцевого пептиду до рекомбінантно продукованої С-кінцевої частини. Як методи сульфуризації / десульфурації, так і використання знімних допоміжних речовин тиолу передбачають встановлення синтетичного тіолового фрагмента для проведення стандартної хімії природного хімічного перев'язування з подальшим видаленням допоміжного / тіольного.
Спрямована еволюція
Основною метою білкової інженерії є розробка нових пептидів або білків з бажаною структурою та хімічною активністю. Оскільки наші знання про зв'язок між первинною послідовністю, структурою та функцією білків обмежені, раціональне проектування нових білків з інженерною діяльністю є надзвичайно складним завданням. При спрямованій еволюції повторні цикли генетичної диверсифікації з подальшим процесом скринінгу або відбору можуть бути використані для імітації природного відбору в лабораторії для проектування нових білків з бажаною активністю.[8]
Існує кілька методів створення великих бібліотек варіантів послідовностей. Серед найбільш широко використовуваних є піддавання ДНК УФ-випромінюванню або хімічним мутагенам, схильній до помилок ПЛР, виродженим кодонам або рекомбінації .[9][10] Після створення великої бібліотеки варіантів використовуються методи вибору або екранування для пошуку мутантів з потрібним атрибутом. Поширені методи відбору / скринінгу включають FACS,[11] дисплей мРНК,[12] фаговий дисплей та <i id="mwdw">in vitro</i> компартменталізацію .[13] Після того, як будуть знайдені корисні варіанти, їх послідовність ДНК ампліфікується і піддається подальшому раунду диверсифікації та відбору.
Нобелівську премію з хімії 2018 року присудили Френсіс Арнольд, Джордж Сміт та Грегорі Вінтер за їх піонерську роботу в напрямку спрямованої еволюції .[14]
Біоортогональні реакції
Для успішного маркування молекули, що цікавить, потрібна спеціальна функціоналізація цієї молекули для хіміоспецифічної реакції з оптичним зондом. Щоб експеримент із маркування вважався надійним, функціоналізація повинна мінімально порушувати систему.[15] На жаль, ці вимоги часто важко виконати. Багато реакцій, зазвичай доступних органічним хімікам в лабораторії, недоступні в живих системах. Водо- і відновно-чутливі реакції не протікали б, реагенти, схильні до нуклеофільної атаки, не забезпечували б хіміоспецифічності, а будь-які реакції з великими кінетичними бар'єрами не знайшли б достатньо енергії у відносно низькому нагріванні середовища живої клітини. Таким чином, хіміки нещодавно розробили групу біоортогональної хімії, яка протікає хіміоспецифічно, незважаючи на середовище відволікаючих реактивних матеріалів in vivo .
З'єднання зонда з цікавою молекулою повинно відбуватися протягом досить короткого проміжку часу; отже, кінетика реакції сполучення повинна бути дуже сприятливою. Хімія клацань добре підходить для заповнення цієї ніші, оскільки реакції клацання швидкі, спонтанні, вибіркові та високопродуктивні.[16] На жаль, найвідоміший "клацання реакції, " а [3 + 2] циклоприєднання між азида і ациклического алкінілу, є мідь-каталізуються, що створює серйозну проблему для використання в природних умовах з — за токсичності Copper.[17] Щоб обійти необхідність в каталізаторі, лабораторія Кероліна Р. Бертоцці ввела притаманний штам алкінових видів за допомогою циклічного алкіну. Зокрема, циклооктин реагує з азидо-молекулами з характерною енергійністю.[18]
Найбільш поширений метод встановлення біоортогональної реактивності в цільовій біомолекулі — це метаболічне маркування. Клітини занурені в середовище, де доступ до поживних речовин обмежений синтетично модифікованими аналогами стандартних видів палива, таких як цукри. Як наслідок, ці змінені біомолекули включаються в клітини так само, як і немодифіковані метаболіти. Потім в систему включається зонд для зображення долі змінених біомолекул. Інші методи функціоналізації включають ферментативне введення азидів у білки[19] та синтез фосфоліпідів, кон'югованих із циклооктинами.[20]
Виявлення біомолекул за допомогою метагеноміки
Досягнення сучасних технологій послідовності в кінці 1990-х років дозволили вченим досліджувати ДНК спільнот організмів у їх природних середовищах («еДНК»), не культивуючи окремих видів у лабораторії. Цей метагеномічний підхід дозволив вченим вивчити широкий вибір організмів, які раніше не характеризувалися, частково через некомпетентні умови росту. Джерела еДНК включають ґрунти, океан, підземну поверхню, гарячі джерела, гідротермальні отвори, полярні крижані шапки, гіперсалінічні середовища існування та екстремальні середовища з рН.[21] З багатьох застосувань метагеноміки такі дослідники, як Джо Хандельсман, Джон Кларді та Роберт М. Гудман, досліджували метагеномічні підходи до відкриття біологічно активних молекул, таких як антибіотики .[22]
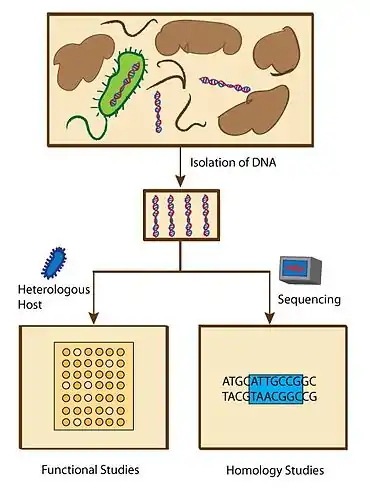
Функціональні або гомологічні скринінг-стратегії використовувались для ідентифікації генів, які виробляють невеликі біоактивні молекули. Функціональні метагеномічні дослідження розроблені для пошуку конкретних фенотипів, які пов'язані з молекулами з певними характеристиками. Метагеномічні дослідження гомології, з іншого боку, призначені для вивчення генів для виявлення збережених послідовностей, які раніше були пов'язані з експресією біологічно активних молекул.[23]
Функціональні метагеномічні дослідження дозволяють виявити нові гени, що кодують біологічно активні молекули. Ці аналізи включають аналізи накладення верхнього агару, де антибіотики генерують зони інгібування росту проти досліджуваних мікробів, і pH-аналізи, які можуть екранувати зміну pH завдяки новосинтезованим молекулам, використовуючи показник рН на планшеті з агаром .[24] Скринінг субпренованої субстратом експресії генів (SIGEX), метод скринінгу експресії генів, які індукуються хімічними сполуками, також був використаний для пошуку генів із специфічними функціями. Метагеномічні дослідження, засновані на гомології, призвели до швидкого виявлення генів, які мають гомологічні послідовності, як відомі раніше гени, відповідальні за біосинтез біологічно активних молекул. Як тільки гени секвенуються, вчені можуть порівнювати тисячі бактеріальних геномів одночасно.[23] Перевага перед функціональними метагеномічними аналізами полягає в тому, що метагеномічні дослідження гомології не потребують системи організму господаря для експресії метагеномів, таким чином цей метод може потенційно заощадити час, витрачений на аналіз нефункціональних геномів. Це також призвело до відкриття кількох нових білків і малих молекул.[25] Крім того, в дослідженні кремнію на основі метагеномічного дослідження Глобального океану було виявлено 20 нових лантибіотичних циклаз .[26]
Посттрансляційна модифікація білків з фосфатними групами кіназами є ключовим регуляторним кроком у всіх біологічних системах. Події фосфорилювання, або фосфорилювання білковими кіназами, або дефосфорилювання фосфатазами, призводять до активації білка або дезактивації. Ці події впливають на регуляцію фізіологічних шляхів, що робить здатність розсікати та вивчати ці шляхи невід'ємною частиною розуміння деталей клітинних процесів. Існує ряд проблем — а саме розмір фосфопротеому, швидкоплинний характер подій фосфорилювання та пов'язані з цим фізичні обмеження класичних біологічних та біохімічних методів — які обмежували просування знань у цій галузі.[27]
Завдяки використанню модуляторів малих молекул білкових кіназ хімічні біологи отримали краще розуміння ефектів фосфорилювання білка. Наприклад, неселективні та селективні інгібітори кінази, такі як клас піридинілімідазолових сполук[28] є потужними інгібіторами, корисними для розсічення сигнальних шляхів кіназної MAP . Ці піридинілімідазольні сполуки функціонують, орієнтуючись на кишеню зв'язування АТФ . Хоча цей підхід, як і пов'язані з цим підходи[29][30] з незначними модифікаціями, виявився ефективним у ряді випадків, ці сполуки не мають достатньої специфіки для більш загальних застосувань. Інший клас сполук, заснований на механізмі інгібіторів, поєднує знання ензимології кінази з раніше використаними мотивами інгібування. Наприклад, «аналог бісубстрату» інгібує дію кінази шляхом зв'язування як збереженого кишені зв'язування АТФ, так і сайту розпізнавання білка / пептиду на специфічній кіназі.[31] Дослідницькі групи також використовували аналоги АТФ як хімічні зонди для вивчення кіназ та ідентифікації їх субстратів.[32][33][34]
Успіхи хімічної біології також покращилися за допомогою класичних методів візуалізації дії кінази. Наприклад, розробка пептидних біосенсорів — пептидів, що містять вбудовані фторофори, покращила тимчасову роздільну здатність тестів на зв'язування in vitro.[35] Однією з найкорисніших методик вивчення дії кінази є флуоресцентна резонансна передача енергії | FRET . Щоб використовувати FRET для досліджень фосфорилювання, флуоресцентні білки з'єднуються як з доменом зв'язування фосфоамінокислоти, так і з пептидом, який може фосфорилюватися. При фосфорилюванні або дефосфорилюванні субстратного пептиду відбувається конформаційна зміна, яка призводить до зміни флуоресценції.[36] FRET також використовувались у тандемі з флуоресцентною довічною мікроскопією візуалізації (FLIM)[37] або флуоресцентно кон'югованими антитілами та проточною цитометрією[38] для отримання кількісних результатів з відмінною часовою та просторовою роздільною здатністю.
Біологічна флуоресценція
Хімічні біологи часто вивчають функції біологічних макромолекул, використовуючи методи флуоресценції . Перевага флуоресценції порівняно з іншими методами полягає у її високій чутливості, неінвазивності, безпечному виявленні та здатності модулювати сигнал флуоресценції. Відкриття рогером Ю. Циєном та іншими особами зеленого флуоресцентного білка (GFP), гібридних систем та квантових точок дозволило більш точно оцінити розташування та функціонування білка.[39] Використовуються три основні типи флюорофорів: невеликі органічні барвники, зелені флуоресцентні білки та квантові крапки . Невеликі органічні барвники зазвичай менше 1 кДа і були модифіковані для підвищення фотостабільності та яскравості та зменшення самозатухання. Квантові точки мають дуже різку довжину хвилі, високу молярну поглинальну здатність та квантовий вихід. Як органічні барвники, так і квантові барвники не мають здатності розпізнавати цікавий білок без допомоги антитіл, отже, вони повинні використовувати імуномарки . Флуоресцентні білки генетично кодуються і можуть бути злиті з вашим білком, який вас цікавить. Ще одна техніка генетичного маркування — тетрацистеїнова біарсенічна система, яка потребує модифікації цільової послідовності, яка включає чотири цистеїни, які зв'язують мембраннопроникні біарсенічні молекули, зелений та червоний барвники «FlAsH» та «ReAsH», з пікомолярною спорідненістю. І флуоресцентні білки, і біарсенічний тетрацистеїн можуть експресуватися в живих клітинах, але вони мають великі обмеження в позаматкової експресії і можуть спричинити втрату функції.
Флуоресцентні методи були використані для оцінки ряду білкової динаміки, включаючи відстеження білка, конформаційні зміни, взаємодія білок-білок, синтез і оборот білка, активність ферментів. Три загальні підходи для вимірювання перерозподілу та дифузії білкової сітки — це відстеження одночастинок, кореляційна спектроскопія та фотомаркирування. Під час відстеження однієї частинки окрема молекула повинна бути достатньо яскравою та розрідженою, щоб відстежуватись від одного відео до іншого. Кореляційна спектроскопія аналізує коливання інтенсивності, що виникають внаслідок міграції флуоресцентних об'єктів у невеликий об'єм та з нього у фокусі лазера. При фотомаркеруванні флуоресцентний білок може бути децензований в субклітинній області із застосуванням інтенсивного локального освітлення, а доля позначеної молекули може бути зображена безпосередньо. Майкалет та його співробітники використовували квантові точки для відстеження одночастинок, використовуючи біотин-квантові точки в клітинах HeLa.[40] Один з найкращих способів виявити конформаційні зміни в білках — це маркування білка, що цікавить, двома фторофорами в безпосередній близькості. FRET відповість на внутрішні конформаційні зміни, що є результатом переорієнтації одного фторофору відносно іншого. Можна також використовувати флуоресценцію для візуалізації активності ферментів, як правило, використовуючи протеоміки на основі загартуваної активності (qABP). Ковалентне зв'язування qABP з активним сайтом цільового ферменту забезпечить прямі докази щодо того, чи відповідає фермент за сигнал після вивільнення гасителя та відновлення флуоресценції.[41]
Див. також
Список літератури
- Modification-specific proteomics: strategies for characterization of post-translational modifications using enrichment techniques. Proteomics 9 (20): 4632–41. October 2009. PMC 2892724. PMID 19743430. doi:10.1002/pmic.200900398. Проігноровано невідомий параметр
|vauthors=(довідка) - Saxon, E. (17 березня 2000). Cell Surface Engineering by a Modified Staudinger Reaction. Science (American Association for the Advancement of Science (AAAS)) 287 (5460): 2007–2010. ISSN 0036-8075. doi:10.1126/science.287.5460.2007.
- Next-Generation Drugs and Probes for Chromatin Biology: From Targeted Protein Degradation to Phase Separation. Molecules 23 (8): 1958. August 2018. PMC 6102721. PMID 30082609. doi:10.3390/molecules23081958. Проігноровано невідомий параметр
|vauthors=(довідка) - Synthesis of proteins by native chemical ligation. Science 266 (5186): 776–779. 1994. Bibcode:1994Sci...266..776D. PMID 7973629. doi:10.1126/science.7973629. Проігноровано невідомий параметр
|vauthors=(довідка) - Expressed protein ligation: a general method for protein engineering. Proc. Natl. Acad. Sci. USA 95 (12): 6705–10. June 1998. Bibcode:1998PNAS...95.6705M. PMC 22605. PMID 9618476. doi:10.1073/pnas.95.12.6705. Проігноровано невідомий параметр
|vauthors=(довідка) - Building complex glycopeptides: Development of a cysteine-free native chemical ligation protocol. Angew. Chem. Int. Ed. Engl. 45 (25): 4116–25. June 2006. PMID 16710874. doi:10.1002/anie.200600538. Проігноровано невідомий параметр
|vauthors=(довідка) - Auxiliary-mediated site-specific peptide ubiquitylation. Angew. Chem. Int. Ed. Engl. 46 (16): 2814–8. 2007. PMID 17366504. doi:10.1002/anie.200605155. Проігноровано невідомий параметр
|vauthors=(довідка) - Protein design by directed evolution. Annu Rev Biophys 37: 153–73. 2008. PMID 18573077. doi:10.1146/annurev.biophys.37.032807.125832. Проігноровано невідомий параметр
|vauthors=(довідка) - Searching sequence space for protein catalysts. Proc. Natl. Acad. Sci. USA 98 (19): 10596–601. September 2001. Bibcode:2001PNAS...9810596T. PMC 58511. PMID 11535813. doi:10.1073/pnas.191159298. Проігноровано невідомий параметр
|vauthors=(довідка) - Directed evolution of protein enzymes using nonhomologous random recombination. Proc. Natl. Acad. Sci. USA 101 (18): 7011–6. May 2004. Bibcode:2004PNAS..101.7011B. PMC 406457. PMID 15118093. doi:10.1073/pnas.0402202101. Проігноровано невідомий параметр
|vauthors=(довідка) - High-throughput screens and selections of enzyme-encoding genes. Curr Opin Chem Biol 9 (2): 210–6. April 2005. PMID 15811807. doi:10.1016/j.cbpa.2005.02.002. Проігноровано невідомий параметр
|vauthors=(довідка) - The use of mRNA display to select high-affinity protein-binding peptides. Proc. Natl. Acad. Sci. USA 98 (7): 3750–5. March 2001. Bibcode:2001PNAS...98.3750W. PMC 31124. PMID 11274392. doi:10.1073/pnas.061028198. Проігноровано невідомий параметр
|vauthors=(довідка) - Man-made cell-like compartments for molecular evolution. Nature Biotechnology 16 (7): 652–6. 1998. PMID 9661199. doi:10.1038/nbt0798-652. Проігноровано невідомий параметр
|vauthors=(довідка) - The Nobel Prize | Women who changed science | Frances H. Arnold. www.nobelprize.org (англ.). Процитовано 7 лютого 2020.
- Bioorthogonal chemistry: fishing for selectivity in a sea of functionality. Angew. Chem. Int. Ed. Engl. 48 (38): 6974–98. 2009. PMC 2864149. PMID 19714693. doi:10.1002/anie.200900942. Проігноровано невідомий параметр
|vauthors=(довідка) - Click Chemistry: Diverse Chemical Function from a Few Good Reactions. Angew. Chem. Int. Ed. Engl. 40 (11): 2004–2021. June 2001. PMID 11433435. doi:10.1002/1521-3773(20010601)40:11<2004::AID-ANIE2004>3.0.CO;2-5. Проігноровано невідомий параметр
|vauthors=(довідка) - A stepwise huisgen cycloaddition process: copper(I)-catalyzed regioselective "ligation" of azides and terminal alkynes. Angew. Chem. Int. Ed. Engl. 41 (14): 2596–9. July 2002. PMID 12203546. doi:10.1002/1521-3773(20020715)41:14<2596::AID-ANIE2596>3.0.CO;2-4. Проігноровано невідомий параметр
|vauthors=(довідка) - A strain-promoted [3 + 2] azide-alkyne cycloaddition for covalent modification of biomolecules in living systems. J. Am. Chem. Soc. 126 (46): 15046–7. November 2004. PMID 15547999. doi:10.1021/ja044996f. Проігноровано невідомий параметр
|vauthors=(довідка) - Crosslinking studies of protein–protein interactions in nonribosomal peptide biosynthesis. Chem. Biol. 16 (4): 372–81. April 2009. PMC 2743379. PMID 19345117. doi:10.1016/j.chembiol.2009.02.009. Проігноровано невідомий параметр
|vauthors=(довідка) - Selective fluorescence labeling of lipids in living cells. Angew. Chem. Int. Ed. Engl. 48 (8): 1498–500. 2009. PMID 19145623. doi:10.1002/anie.200805507. Проігноровано невідомий параметр
|vauthors=(довідка) - Tapping into microbial diversity. Nature Reviews Microbiology 2 (2): 141–50. February 2004. PMID 15040261. doi:10.1038/nrmicro819. Проігноровано невідомий параметр
|vauthors=(довідка) - Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products. Chem. Biol. 5 (10): R245–9. October 1998. PMID 9818143. doi:10.1016/S1074-5521(98)90108-9. Проігноровано невідомий параметр
|vauthors=(довідка); Проігноровано невідомий параметр|doi-access=(довідка) - Recent application of metagenomic approaches toward the discovery of antimicrobials and other bioactive small molecules. Current Opinion in Microbiology 13 (5): 603–609. 2010. PMC 3111150. PMID 20884282. doi:10.1016/j.mib.2010.08.012. Проігноровано невідомий параметр
|vauthors=(довідка) - Daniel R (2005). The metagenomics of soil. Nature Reviews Microbiology 3 (6): 470–478. PMID 15931165. doi:10.1038/nrmicro1160.
- Identification and characterization of lipolytic enzymes from a peat-swamp forest soil metagenome. Biosci. Biotechnol. Biochem. 74 (9): 1848–54. 2010. PMID 20834152. doi:10.1271/bbb.100249. Проігноровано невідомий параметр
|vauthors=(довідка); Проігноровано невідомий параметр|doi-access=(довідка) - Catalytic promiscuity in the biosynthesis of cyclic peptide secondary metabolites in planktonic marine cyanobacteria. Proceedings of the National Academy of Sciences of the United States of America 107 (23): 10430–10435. 2010. Bibcode:2010PNAS..10710430L. PMC 2890784. PMID 20479271. doi:10.1073/pnas.0913677107. Проігноровано невідомий параметр
|vauthors=(довідка) - The Chemical Biology of Protein Phosphorylation. Annual Review of Biochemistry 78: 797–825. 2009. PMC 3074175. PMID 19489734. doi:10.1146/annurev.biochem.78.070907.103047. Проігноровано невідомий параметр
|vauthors=(довідка) - The structural basis for the specificity of pyridinylimidazole inhibitors of p38 MAP kinase. Chemistry & Biology 4 (6): 423–31. June 1997. PMID 9224565. doi:10.1016/S1074-5521(97)90194-0. Проігноровано невідомий параметр
|vauthors=(довідка); Проігноровано невідомий параметр|doi-access=(довідка) - Inhibition of p38 MAP kinase by utilizing a novel allosteric binding site. Nature Structural Biology 9 (4): 268–72. April 2002. PMID 11896401. doi:10.1038/nsb770. Проігноровано невідомий параметр
|vauthors=(довідка) - Structural mechanism for STI-571 inhibition of abelson tyrosine kinase. Science 289 (5486): 1938–42. September 2000. Bibcode:2000Sci...289.1938S. PMID 10988075. doi:10.1126/science.289.5486.1938. Проігноровано невідомий параметр
|vauthors=(довідка) - Mechanism-based design of a protein kinase inhibitor. Nature Structural Biology 8 (1): 37–41. January 2001. PMID 11135668. doi:10.1038/83028. Проігноровано невідомий параметр
|vauthors=(довідка) - A Cell-Permeable ATP Analogue for Kinase-Catalyzed Biotinylation. Angewandte Chemie 54 (33): 9618–21. August 2015. PMC 4551444. PMID 26119262. doi:10.1002/anie.201503041. Проігноровано невідомий параметр
|vauthors=(довідка) - The generality of kinase-catalyzed biotinylation. Bioorganic & Medicinal Chemistry 24 (1): 12–9. January 2016. PMC 4921744. PMID 26672511. doi:10.1016/j.bmc.2015.11.029. Проігноровано невідомий параметр
|vauthors=(довідка) - Anthony, Thilani M.; Dedigama-Arachchige, Pavithra M.; Embogama, D. Maheeka; Faner, Todd R.; Fouda, Ahmed E.; Pflum, Mary Kay H. (2015). ATP Analogs in Protein Kinase Research. У Kraatz, Heinz-Bernhard; Martic, Sanela. Kinomics: Approaches and Applications. с. 137–68. ISBN 978-3-527-68303-1. doi:10.1002/9783527683031.ch6.
- Peptide-based fluorescent sensors of protein kinase activity: design and applications. Biochim. Biophys. Acta 1784 (1): 94–9. January 2008. PMC 2684651. PMID 17881302. doi:10.1016/j.bbapap.2007.07.016. Проігноровано невідомий параметр
|vauthors=(довідка) - A genetically encoded fluorescent reporter reveals oscillatory phosphorylation by protein kinase C. J. Cell Biol. 161 (5): 899–909. 2003. PMC 2172956. PMID 12782683. doi:10.1083/jcb.200302125. Проігноровано невідомий параметр
|vauthors=(довідка) - Quantitative imaging of lateral ERbB1 receptor signal propagation in the plasma membrane. Science 290 (5496): 1567–1570. 2000. Bibcode:2000Sci...290.1567V. PMID 11090353. doi:10.1126/science.290.5496.1567. Проігноровано невідомий параметр
|vauthors=(довідка) - Kinetics and extent of protein tyrosine kinase activation in individual T cells upon antigenic stimulation. Immunology 97 (2): 287–293. 1999. PMC 2326824. PMID 10447744. doi:10.1046/j.1365-2567.1999.00767.x. Проігноровано невідомий параметр
|vauthors=(довідка) - The Fluorescent Toolbox for Assessing Protein Location and Function. Science 312 (5771): 217–224. 2006. Bibcode:2006Sci...312..217G. PMID 16614209. doi:10.1126/science.1124618. Проігноровано невідомий параметр
|vauthors=(довідка) - Quantum Dots for Live Cells, in Vivo Imaging, and Diagnostics. Science 307 (5709): 538–544. 2005. Bibcode:2005Sci...307..538M. PMC 1201471. PMID 15681376. doi:10.1126/science.1104274. Проігноровано невідомий параметр
|vauthors=(довідка) - Fluorescent probes for bioimaging applications. Current Opinion in Chemical Biology 12 (5): 515–21. 2008. PMID 18771748. doi:10.1016/j.cbpa.2008.08.007. Проігноровано невідомий параметр
|vauthors=(довідка)
Подальше читання
Журнали
- ACS Chemical Biology — новий журнал хімічної біології Американського хімічного товариства.
- Біоорганічна та лікарська хімія — Журнал тетраедрів для досліджень на стику хімії та біології
- ChemBioChem — Європейський журнал хімічної біології
- Хімічна біологія — точка доступу до новин хімічної біології та досліджень з усіх публікацій RSC
- Cell Chemical Biology — Міждисциплінарний журнал, який публікує праці, що представляють винятковий інтерес у всіх областях на межі між хімією та біологією. chembiol.com
- Журнал хімічної біології — новий журнал, що публікує нову працю та огляди на межі між біологією та фізичними науками, опублікований Springer. посилання
- Journal of the Royal Society Interface — міждисциплінарна публікація, що сприяє дослідженню на межі фізичних та життєвих наук
- Molecular BioSystems — журнал хімічної біології з особливим акцентом на взаємозв'язок між хімією та -омічними науками та системною біологією.
- Nature Chemical Biology — щомісячний мультидисциплінарний журнал, що надає міжнародний форум для своєчасного опублікування значних нових досліджень на межі між хімією та біологією.
- Wiley Енциклопедія хімічної біології посилання