5S рРНК
5S рибосомна РНК (5S рРНК) — молекула рибосомної РНК що має довжину 120 нт і молекулярну масу близько 40000 Da. Вона є структурним і функціональним компонентом великих рибосомних субодиниць в основних доменах живого (бактерії, археї, еукаріоти), за виключенням мітохондріальних рибосом у грибів і тварин. Позначення 5S походить від швидкості седиментації цієї молекули під час ультрацентригування у одиницях Сведберга (S).[1]
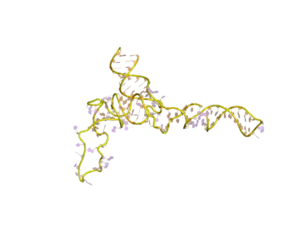
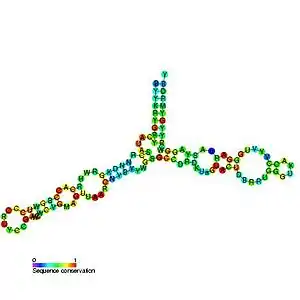
Біосинтез
У прокаріотів, ген 5S рРНК розташований в опероні рРНК після генів що кодують малу і велику субодиницю рРНК. Ці гени ко-транскрибуються в один поліцистронний прекурсор.[3] Особливістю еукаріотичних ядерних геномів є наявність багатьох копій гену 5S рРНК (5S рДНК), які згруповані у тандемні повтори. Кількість копій варіюється у різних видів.[4][5] Еукаріотична 5S рРНК синтезується РНК-полімеразою III, тоді як інші еукаріотичні рРНК утворюється в процесі розрізання 45S прекурсору, який був транскрибований РНК-полімеразою I. В ооцитах Xenopus було показано, що домени типу «цинковий палець» 4-7 фактора транскрипції TFIIIA можуть зв'язуватися з центральною ділянкою 5S рРНК.[6][7] Взаємодія між 5S рРНК і TFIIIA одночасто зменшує транскрипцію генів 5S рРНК, а також стабілізує вже синтезовані транскрипти 5S рРНК до того моменту коли вони будуть використані для збірки рибосом.[8]
Структура
Вторинна структура 5S рРНК представлена п'ятьма спіральними ділянками (позначаються римськими цифрами I—V), чотирма петлями (B-E), та однією рухомою петлею (A), які всі разом утворюють Y-подібну тривимірну структуру. Петлі C і D розташовані на периферії, тоді як петлі B і E є внутрішніми.[4] Філогенетичні дослідження вказують на те що спіралі I та III є спорідненими.[9] Спіраль III містить два висококонсервативних аденозини.[10] Спіраль V, що містить шпилькоподібну структуру, бере участь у взаємодії з TFIIIA.[4]
Розташування в контексті рибосоми
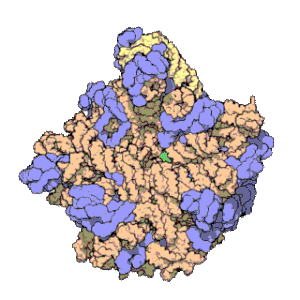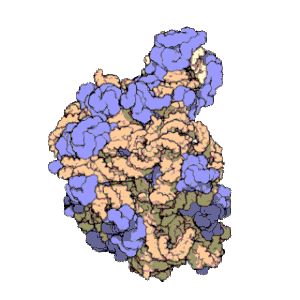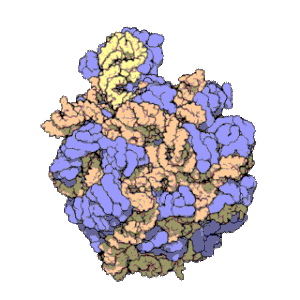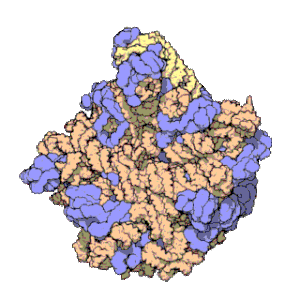
За допомогою різноманітний методів, таких як кріо-електронна мікроскопія, хімічний кросс-лінкінг, а також рентгеноструктурний аналіз, фізичне розташування 5S рРНК в структурному контексті рибосоми було визначене з великою точністю. У бактерій та архей, велика рибосомна субодиниця складається з двох молекул РНК, 5S рРНК, а також іншої, більшої молекули РНК, відомої як 23S рРНК, які асоційовані з численними рибосомними білками.[3] У еукаріотів, велика рибосомна субодиниця містить 5S, 5.8S та 28S рРНК, а також велику кількість молекул білків.[12][13] Тривимірна будова великої субодиниці утворює відносно однорідну молекулярну поверхню, на якій виділяють три помітні топологічні елементи: протруберанець L1, центральний протруберанець, а також стебло L7/L12. Протруберанець L1 та стебло L7/L12 розташовані по боках центрального протруберанцю. 5S рРНК розташована в центральному протруберанці. Іншими складовими центрального протруберанцю є 23S рРНК (28S рРНК в еукаріотів) а також рибосомні білки L5, L18, L25, L27.[14]
Функціональна роль в рибосомі
Функції 5S рРНК вивчені ще не повнівстю. В Escherichia coli, делеція генів 5S рРНК знижує швидкість біосинтезу білків а також має більш виражений загальний негативний вплив на функціонування бактеріальної клітини ніж аналогічні делеції ідентичної кількості генів 16S і 23S рРНК.[15] Кристалографічні дослідження вказують на те що 5S-рРНК-зв'язуючі білки а також інші білки у складі великого протруберанцю великої рибосомної субодиниці грають роль у взаємодії рибосоми з тРНК.[14] Окрім цього, переплетення 5S рРНК і 23S рРНК, завдяки яким утворюються пептидилтрансферазний і ГТФазний сайти, свідчать про те що 5S рРНК діє як медіатор між двома функціональними сайтами рибосоми, приймаючи роль у взаємодії між двома рибосомними субодиницями і взаємодіях рибосоми з тРНК.[14]
Роль в біогенезі рибосоми
У еукаріотів, цитоплазматичні рибосоми формуються з чотирьох рибосомних РНК та більш ніж 80 рибосомних білків.[13][16] Після транскрипції, 3’ кінці 5S рРНК обробляються екзонуклеазами Rex1p, Rex2p, і Rex3p.[4] Субодиниці 60S та 40S експортуються з ядра в цитоплазму, де процес формування трансляційно-компетентної 80S рибосоми завершується. Точний етап біогенезу рибосоми під час якого відбувається вбудовування 5S рРНК залишається невизначеними,[4] але консесусною є точка зору, що 5S рРНК входить до складу 90S-частинки, яка є прекурсором 60S субодиниці; при цьому 5S рРНК існує у вигляді малого рибонуклеопротеїнового комплексу з рибосомним білком L5.[16]
Взаємодія з білками
Білок La
Взаємодія між 5S рРНК і білком La захищає РНК від екзонуклеазної деградації в клітині.[17] Білок La знайдений в ядрах всіх еукаріотичних організмів і асоціюється з декількома типами РНК які транскрибуються РНК-полімеразою III. Білок La взаємодіє з РНК (включаючи 5S рРНК) через 3’ олігоурідиновий фрагмент, сприяючи стабілізації РНК.[4][18]
Рибосомний білок L5
В еукаріотичних клітинах, рибосомний білок L5 асоціюється з 5S рРНК, утворюючи стабільну прерибосомну рибонуклеопротеїнову частинку, яка детектується і в цитозолі, і в ядрі. Дефіцит L5 зашкоджає транспорту 5S рРНК в ядро і негативно впливає на процес збірки рибосом.[4]
Інші рибосомні білки
У прокаріотів 5S рРНК взаємодіє з рибосомними білками L5, L18 і L25. В еукаріотів єдиним відомим партнером 5S рРНК є рибосомний білок L5.[19] У T. brucei, що викликає сонну хворобу, 5S рРНК взаємодіє з двома спорідненими білками P34 та P37; втрата цих білків призводить до зменшення рівня 5S рРНК в клітині.[4]
Альтернативні 5S рРНК в рибосомах органел
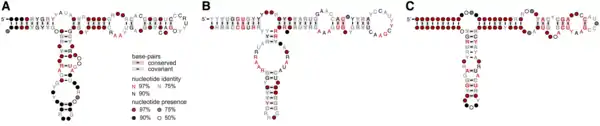
Трансляційний апарат мітохондрій і пластид (органели ендосимбіотичного бактеріального походження), порівняно з трансляційним апаратом бактерій, має багато спільних рис, але одночасно з цим має і суттєві відмінності. Геноми всіх органел без виключень кодують велику і малу субодиниці рибосоми, в той же час розповсюдження гену 5S рРНК (rrn5) є менш універсальним у різних організмів. Rrn5 присутній у геномах всіх пластид. Мітохондріальний rrn5 є тільки у рослин і малої кількості протистів.[20][21]
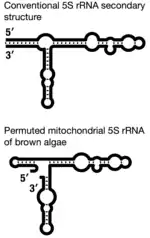
Мітохондіральна 5S рРНК більшості гетероконтів демонструє широке розмаїття вторинних структур.[22] Найбільш незвичною є мітохондріальна 5S рРНК з бурих водоростей, в якої закриваюча спіраль I, яка мала б поєднувати 5′ і 3′ кінці молекули, замінена на шпильку (малюнок).
Мітохондріальні геноми тварин, грибів, авльвеолобіонтів і евгленових не мають гену 5S рРНК.[22] Центральний протруберанець в рибосомах цих організмів перебудований декількома способами. В грибних рибосомах, 5S рРНК замінена подовженою рРНК великої рибосомної субодиниці.[23] В кінетопластидах, центральний протруберанець мітохондріальних рибосом сформований еволюційно новими рибосомними білками.[24] Нарешті, мітохондріальні рибосоми тварин прилаштували спеціальну рибосомну тРНК на місце відсутньої 5S рРНК.[25][26]
Див. також
- 50S рРНК
- Трансляція
- Рибосома
Посилання
- 5S Ribosomal RNA Database. Nucleic Acids Res. 30 (1). January 2002. с. 176–8. PMC 99124. PMID 11752286. doi:10.1093/nar/30.1.176.
- The 3D arrangement of the 23 S and 5 S rRNA in the Escherichia coli 50 S ribosomal subunit based on a cryo-electron microscopic reconstruction at 7.5 A resolution.. J Mol Biol 298 (1). 2000. с. 35–59. PMID 10756104. doi:10.1006/jmbi.2000.3635.
- Kaczanowska, M; Rydén-Aulin, M (September 2007). Ribosome biogenesis and the translation process in Escherichia coli.. Microbiology and molecular biology reviews 71 (3). с. 477–494. PMID 17804668. doi:10.1128/MMBR.00013-07.
- Ciganda, Martin; Williams, Noreen (July 2011). Eukaryotic 5S rRNA biogenesis. Wiley Interdisciplinary Reviews: RNA 2 (4). с. 526. doi:10.1002/wrna.74.
- Douet, J; Tourmente, S (Jul 2007). Transcription of the 5S rRNA heterochromatic genes is epigenetically controlled in Arabidopsis thaliana and Xenopus laevis.. Heredity 99 (1). с. 5–13. PMID 17487217. doi:10.1038/sj.hdy.6800964.
- McBryant, SJ; Veldhoen, N; Gedulin, B; Leresche, A; Foster, MP; Wright, PE; Romaniuk, PJ; Gottesfeld, JM (1995). Interaction of the RNA binding fingers of Xenopus transcription factor IIIA with specific regions of 5 S ribosomal RNA.. Journal of Molecular Biology 248 (1). с. 44–57. PMID 7731045. doi:10.1006/jmbi.1995.0201.
- Searles, MA; Lu D; Klug A (2000). The role of the central zinc fingers of transcription factor IIIA in binding to 5 S RNA. J Mol Biol 301 (1). с. 47–60. PMID 10926492. doi:10.1006/jmbi.2000.3946.
- Pelham, HRB; Brown DD (1980). A specific transcription factor that can bind either the 5S RNA gene or 5S RNA. Proc. Natl. Acad. Sci. USA 77 (7). с. 4170–4174. PMC 349792. PMID 7001457. doi:10.1073/pnas.77.7.4170.
- Sun, FJ; Caetano-Anollés, G (Nov 2009). The evolutionary history of the structure of 5S ribosomal RNA.. Journal of Molecular Evolution 69 (5). с. 430–43. PMID 19639237. doi:10.1007/s00239-009-9264-z.
- DiNitto, JP; Huber, PW (23 жовтня 2001). A role for aromatic amino acids in the binding of Xenopus ribosomal protein L5 to 5S rRNA.. Biochemistry 40 (42). с. 12645–53. PMID 11601989. doi:10.1021/bi011439m.
- The complete atomic structure of the large ribosomal subunit at 2.4 Å resolution. Science 289 (5481). 2000. с. 905–20. PMID 10937989. doi:10.1126/science.289.5481.905.
- Turowski, TW; Tollervey, D (2015). Cotranscriptional events in eukaryotic ribosome synthesis. Wiley interdisciplinary reviews. RNA 6 (1). с. 129–139. PMID 25176256. doi:10.1002/wrna.1263.
- Yusupova, G; Yusupov, M (February 2014). High-resolution structure of the eukaryotic 80S ribosome.. Annual Review of Biochemistry 83. с. 467–486. PMID 24580643. doi:10.1146/annurev-biochem-060713-035445.
- Gongadze, G. M. (7 січня 2012). 5S rRNA and ribosome. Biochemistry (Moscow) 76 (13). с. 1450–1464. doi:10.1134/S0006297911130062.
- Ammons, D; Rampersad, J; Fox, GE (15 січня 1999). 5S rRNA gene deletions cause an unexpectedly high fitness loss in Escherichia coli.. Nucleic Acids Research 27 (2). с. 637–42. PMC 148226. PMID 9862991. doi:10.1093/nar/27.2.637.
- Henras, AK; Soudet, J; Gérus, M; Lebaron, S; Caizergues-Ferrer, M; Mougin, A; Henry, Y (Aug 2008). The post-transcriptional steps of eukaryotic ribosome biogenesis.. Cellular and molecular life sciences : CMLS 65 (15). с. 2334–59. PMID 18408888. doi:10.1007/s00018-008-8027-0.
- Wolin, SL; Cedervall, T (2002). The La protein.. Annual Review of Biochemistry 71. с. 375–403. PMID 12045101. doi:10.1146/annurev.biochem.71.090501.150003.
- Maraia, RJ; Intine, RV (2002). La protein and its associated small nuclear and nucleolar precursor RNAs.. Gene expression 10 (1-2). с. 41–57. PMID 11868987.
- Moore PB (March 2001). The ribosome at atomic resolution. Biochemistry 40 (11). с. 3243–50. PMID 11258942. doi:10.1021/bi0029402.
- Bullerwell, CE; Schnare, MN; Gray, MW (March 2003). Discovery and characterization of Acanthamoeba castellanii mitochondrial 5S rRNA.. RNA 9 (3). с. 287–292. PMID 12592002. doi:10.1261/rna.2170803.
- Bullerwell, CE; Burger, G; Gott, JM; Kourennaia, O; Schnare, MN; Gray, MW (May 2010). Abundant 5S rRNA-like transcripts encoded by the mitochondrial genome in Amoebozoa.. Eukaryot Cell 9 (5). с. 762–773. PMID 20304999. doi:10.1128/EC.00013-10.
- Valach, M; Burger, G; Gray, MW; Lang, BF (Dec 2014). Widespread occurrence of organelle genome-encoded 5S rRNAs including permuted molecules.. Nucleic Acids Res. 42 (22). с. 13764–13777. PMID 25429974. doi:10.1093/nar/gku1266.
- Amunts, A; Brown, A; Bai, XC; Llácer, JL; Hussain, T; Emsley, P; Long, F; Murshudov, G та ін. (March 2014). Structure of the yeast mitochondrial large ribosomal subunit.. Science 343 (6178). с. 1485–1489. PMID 24675956. doi:10.1126/science.1249410.
- Sharma, MR; Booth, TM; Simpson, L; Maslov, DA; Agrawal, RK (Jun 2009). Structure of a mitochondrial ribosome with minimal RNA.. Proc Natl Acad Sci U S A 106 (24). с. 9637–9642. PMID 19497863. doi:10.1073/pnas.0901631106.
- Brown, A; Amunts, A; Bai, XC; Sugimoto, Y; Edwards, PC; Murshudov, G; Scheres, SH; Ramakrishnan, V (Nov 2014). Structure of the large ribosomal subunit from human mitochondria.. Science 346 (6210). с. 718–722. PMID 25278503. doi:10.1126/science.1258026.
- Greber, BJ; Boehringer, D; Leibundgut, M; Bieri, P; Leitner, A; Schmitz, N; Aebersold, R; Ban, N (Nov 2014). The complete structure of the large subunit of the mammalian mitochondrial ribosome.. Nature 515 (7526). с. 283–286. PMID 25271403. doi:10.1038/nature13895.
