Методи фіксації конформації хромосом
Методи фіксації конформації хромосом, часто скорочено до 3С-технологій або методів на основі 3С (англ. Chromosome conformation capture, 3C)[1] — це набір методів молекулярної біології, що використовуються для аналізу просторової організації хроматину в ядрі клітини. Їх застосовують для кількісної оцінки взаємодій між геномними локусами, які розташовані поблизу в тривимірному просторі, але можуть бути розділені багатьма нуклеотидами в лінійному геномі.[2] Такі взаємодії можуть виникати внаслідок біологічних функцій, наприклад, між промотором і енхансером або в результаті випадкового утворення петель полімеру, коли непрямий фізичний рух хроматину викликає «зіткнення» локусів.[3] При цьому регуляторні елементи можуть розташовуватися на відстані декількох мільйонів пар основ від генів, експресію яких вони контролюють.[4] Незважаючи на це, складна конформація ділянки ДНК між ними дозволяє їм безпосередньо взаємодіяти один з одним.[5]
Частоти взаємодій можуть бути проаналізовані безпосередньо[6] або конвертовані у відстані та використані для реконструкції 3D структур.[7]
Основні відмінності методів на основі 3C — це їх можливості і область застосування.[8] Наприклад, при використанні ПЛР для виявлення взаємодії в експерименті 3С кількісно оцінюються взаємодії двох конкретних фрагментів. Навпаки, Hi-C кількісно визначає взаємодію між усіма можливими парами фрагментів одночасно. Глибоке секвенування матеріалу, отриманого за допомогою 3C, також дозволяє скласти карти взаємодій цілого генома.
Історія
Історично мікроскопія була основним методом дослідження ядерної організації[9], який можна датувати ще 1590 роком.[10]
- У 1879 році Вальтер Флеммінг ввів термін «хроматин»;[11]
- У 1883 році Август Вейсман пов'язав хроматин зі спадковістю;
- У 1884 році Альбрехт Коссель відкрив гістони;
- У 1888 році Саттон і Бовері запропонували теорію безперервності хроматину протягом клітинного циклу;[12]
- У 1889 році Вільгельм фон Вальдейер ввів термін «хромосома»;[13]
- У 1928 році Еміль Хайц ввів термін «гетерохроматин» та «еухроматин»;[14]
- У 1942 році Конрад Воддінгтон постулював епігенетичні ландшафти;[15]
- У 1948 році Роллін Хотчкіс відкрив метилювання ДНК;[16]
- У 1953 році Уотсон і Крік відкрили подвійну спіральну структуру ДНК;[17]
- У 1961 році Мері Лайон постулювала принцип X-інактивації;
- У 1973/1974 роках було виявлено хроматинове волокно;[15]
- У 1975 році П'єр Шамбон ввів термін «нуклеосома»;[15]
- У 1982 році були виявлені хромосомні території;[18]
- У 1984 році Джон Т. Ліс запровадив методику імунопреципітації хроматину;
- У 1993 році був опублікований аналіз ядерного лігування - метод, який міг визначити частоту циркуляції ДНК у розчині. Цей аналіз був використаний, щоб показати, що естроген індукує взаємодію між промотором гена пролактину та сусіднім енхансером;[19]
- У 2002 році Джоб Деккер представив нову ідею про те, що щільні матриці частот взаємодії між локусами можуть використовуватися для виведення просторової організації геномів. Ця ідея стала основою для його розробки аналізу фіксації хромосомної конформації (3С), опублікованого у 2002 році Джобом Деккером та його колегами в лабораторії Клекнера Гарвардського університету;[20][21]
- У 2003 році проект «Геном людини» був закінчений;
- У 2006 році Маріке Сімоні винайшла 4C,[22] а Дості в лабораторії Деккера винайшов 5С;[23]
- У 2007 році Б. Франклін П'ю вніс інновації у методику ChIP-seq;[24]
- У 2009 році Ліберман-Ейден і Джоб Деккер винайшли Hi-C,[25] Меліса Дж. Фулвуд та Іджун Руан винайшли ChIA-PET;[26]
- У 2012 році група під керівництвом Рен та групи під керівництвом Едіт Херд та Джоба Деккера виявили топологічно асоційовані домени (англ. TAD — Topologically Associating Domains) у ссавців;[27][28]
- У 2013 році Такаші Нагано та Пітер Фрейзер ввели методику внутрішньоядерного лігування для Hi-C та одноклітинного Hi-C.[29]
Експериментальні методи
Всі методи фіксації конформації хромосом починаються з аналогічного набору маніпуляцій на початковому етапі, що виконується на зразку клітин:
- Обробка формальдегідом[30], який зшиває ділянки геному, які розташовані поруч у просторі, таким чином заморожуючи взаємодії між локусами. Найчастіше використовується 1-3 % розчин формальдегіду для фіксації протягом 10-30 хвилин при кімнатній температурі.[31] Однак, необхідна стандартизація для запобігання сшивок білків з ДНК, які можуть заважати рестрикції на наступному етапі;
- Фрагментація за допомогою ендонуклеаз рестрикції (рестриктаз). Розмір фрагментів рестрикції визначає роздільну здатність карти взаємодій. Для цього використовуються рестрикційні ферменти (англ. REs — restriction enzymes), які роблять надрізи на послідовності розпізнавання 6 п.н., такі як EcoR1 або HindIII, оскільки вони розрізають геном раз на 4000 п.н., даючи близько 1 мільйона фрагментів у випадку генома людини.[31][32] Для більш точного відображення взаємодій також можуть бути використані 4 п.н. розпізнаваючі рестриктази;
- Випадкове лігування, яке проводиться при низьких концентраціях ДНК в присутності Т4 ДНК-лігази[33] або всередині неушкоджених, пермеабілізованих ядер.[29] В результаті «склеюються» кінці зшитих ДНК. Низькі концентрації забезпечують специфічність лігування (тільки між зшитими взаємодіючими фрагментами). Згодом взаємодіючі локуси кількісно оцінюють шляхом ампліфікації лігованих фрагментів за допомогою ПЛР;[31][33]
- Створення парної 3С бібліотеки. Термічна обробка призводить до розриву зв'язків і утворення лінійних химерних фрагментів ДНК. В результаті буде створена бібліотека взаємодіючих фрагментів ДНК (3C бібліотека);[34]
- Real-time ПЛР дозволяє оцінювати вірогідність взаємодії двох конкретних ділянок геному. Проводять підбір праймерів таким чином, що кожен праймер комплементарний своєму відповідному локусу. У разі взаємодії відбувається відпал обох праймерів і ампліфікація фрагмента.[8]
Базові методи
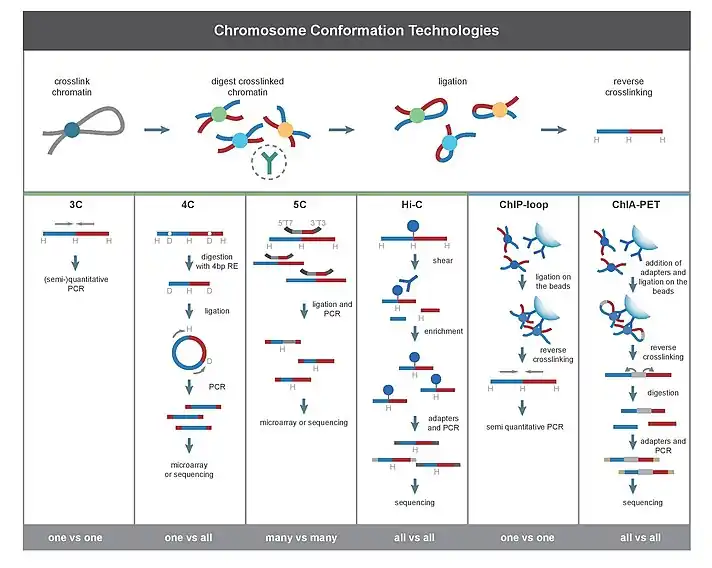
3C (один проти одного)
Метод захоплення конформації хромосоми (англ. Chromosome conformation capture, 3C) необхідний для кількісного визначення взаємодії між обраної парою геномних локусів. Наприклад, 3C можна використовувати для вивчення потенційної взаємодії промотор-енхансер. Ліговані фрагменти детектують за допомогою ПЛР, використовуючи праймери до відомих послідовностей.[10][20] Ось чому ця методика вимагає попередніх знань про взаємодіючі ділянки.
4С (один проти всіх)
Метод замкнутого захоплення конформації хромосоми (англ. Circularized chromosome conformation capture, 4С) охоплює взаємодії між одним обраним локусом і іншими геномними локусами. Він використовується для того, щоб знайти ділянку генома, яка взаємодіє з даною послідовністю ДНК[35] і являє собою комбінацію стандартного методу 3С з інвертованою ПЛР.
Перші 4 етапу збігаються з етапами методу фіксації конформації хромосом. Далі послідовно проводяться фрагментація отриманої 3С бібліотеки рестриктазами; лігування для циклізації фрагментів ДНК, в результаті отримуючи бібліотеку «кільцевих» химерних ДНК (4C бібліотека).[36] Інвертована ПЛР дозволяє ампліфікувати невідому послідовність, використовуючи відому послідовність, зшиту з нею.[22] Аналіз 4С бібліотеки проводиться з використанням ДНК-мікрочипів.
На відміну від 3C і 5C, методи 4C не вимагають попереднього знання нуклеотидних послідовностей обох взаємодіючих хромосомних ділянок.[8] Результати, отримані за допомогою 4С, є надзвичайно відтворюваними при більшості взаємодій, виявлених між регіонами, що є близькими один до одного. На одному мікрочипі можна проаналізувати близько мільйона взаємодій.[1]
5C (багато проти багатьох)
5C розпізнає взаємодії між усіма фрагментами в заданому регіоні, причому розмір цього регіону зазвичай не перевищує мегабази. Він дозволяє шукати ділянки ДНК, які взаємодіють з декількома вибраними ділянками генома і являє собою комбінацію методу 3С і мультиплексной ПЛР.[23]
Перші 4 етапу збігаються з етапами методу фіксації конформації хромосом. Далі послідовно проводяться лігування адаптерів до всіх фрагментів з використанням Taq-лігази; аналіз 5С бібліотеки проводиться з використанням ДНК-мікрочипів і секвенування.[34] 5С корисний для вивчення складних взаємодій, проте має відносно низький покриття. Метод не підходить для вивчення комплексних взаємодій по всьому геному, оскільки для цього будуть потрібні мільйони праймерів 5C.[8]
Hi-C (всі проти всіх)
Hi-C використовує високопродуктивне секвенування для пошуку нуклеотидної послідовності фрагментів.[25] В даному випадку проводиться обробка рестриктазами фіксованого хроматину, біотінілювання кінців і подальше випадкове лігування. Пара послідовностей незалежно картується на геном, що дозволяє виділити фрагменти, задіяні в лігуванні. Таким чином, перевіряються всі можливі парні взаємодії між фрагментами.[29]
Вчені намагаються встановити межі застосування методу Hi-C на прикладі дослідження, присвяченого скринінгу первинних пухлин головного мозку.[37] До онкоскринінгу Hi-C в основному використовувався для досліджень на культурах клітин.[38]
Спеціальні методи
До спеціальних методів можна віднести методи на основі захоплення послідовності, single-cell методи і методи на основі імунопреципітації. Наприклад, single-cell Hi-C може бути використаний для вивчення взаємодій в окремих клітинах.[29]
Методи на основі захоплення послідовності використовують фіксацію олігонуклеотидів для збагачення бібліотек 3C і Hi-C досліджуваних локусів.[39] До них належать: Capture-C[40], NG Capture-C[41], Capture-3C[42] і Capture Hi-C[43]. Ці методи дозволяють досягти більш високої роздільної здатності та чутливості, ніж методи на основі 4C.[8]
Методи на основі імунопреципітації дозволяють виділити локуси, які взаємодіють за допомогою специфічних білків, наприклад, транскрипційних факторів або інсуляторних білків.[44] Серед них можна виділити такі методи, як ChIP-loop і ChIA-PET. ChIP-loop комбінує 3C з ChIP-seq для детекції взаємодії між двома важливими локусами, що опосередкована досліджуваним білком.[45] ChIA-PET поєднує Hi-C та ChIP-seq для детекції всіх взаємодій, опосередкованих досліджуваним білком.[2][26] HiChIP був розроблений з метою аналогічного аналізу, як ChIA-PET з меншою кількістю вхідного матеріалу.[46]
Біологічне значення
Методи 3C сприяли великому числу важливих біологічних відкриттів, включаючи нові дані про структурні особливості хромосом, класифікацію хроматинових петель, а також допомогли поглибити знання про механізми регуляції транскрипції (чиє порушення може призводити до цілого ряду захворювань).[9]
Методи фіксації конформації хромосом продемонстрували важливість просторової близькості регуляторних елементів генів. Наприклад, в тканинах, що експресують гени глобіну, контрольна ділянка локусу β-глобіну формує петлю разом з даними генами. При цьому петля відсутня в тканинах, де цей ген не експресується.[47] Ця технологія надалі допомогла генетичному та епігенетичному вивченню хромосом як у модельних організмів, так і у людини.
Фіксація конформації хромосом дозволила виявити крупний рівень їх організації — так звані ТАДи (топологічно асоційовані домени), що корелюють зі змінами в епігенетичних маркерах. Деякі ТАДи не виявляють транскрипційної активності, в той час як активність інших інгібується.[48] Велика кількість ТАДів виявлено у D. melanogaster, миші і людини.[49] Основну роль у визначенні взаємодій між ТАДами, енхансером і промоторами грають транскрипційні фактор CTCF і білковий комплекс когезин. Результати 3C експериментів говорять про важливість орієнтації («обличчям до обличчя») зв'язуючих мотивів CTCF і енхансер-промоторної петлі. Це необхідно для коректного позиціонування енхансера щодо своєї мішені.[50]
Захворювання людини
Існує ряд хвороб, що викликаються дефектами промотор-енхансерної взаємодії.[51] До них належить таке захворювання крові, як бета-таласемія, що виникає внаслідок делеції енхансерного елемента ЛКО.[52][53] Мутація в SBE2 енхансері, яка, в свою чергу, послаблює експресію гена SHH[54], призводить до розвитку голопрозенцефалії. При цьому порушується формування кінцевого мозку, розділеного на півкулі. Іншим прикладом пов'язаних зі зміною експресії SHH захворювань є полідактилія другого типу PPD2 (трифаланговий великий палець). Вона виникає через мутації регуляторного елемента ZRS, що впливає на посилену продукцію SHH.[55]
Розлад взаємодій між промотором і енхансером впливає не тільки на вади розвитку, але також може служити причиною онкологічних захворювань. Так, аденокарцинома легень може розвиватися внаслідок дуплікації енхансерного елемента гена MYC.[56] T-клітинний гострий лімфобластний лейкоз може бути викликаний появою нового енхансера через мутації в послідовності інтрона.[57]
Аналіз даних
Дані, що виходять в результаті різних 3C експериментів, характеризуються відмінними структурою і статистичними властивостями. Тому для обробки кожного типу експериментів існує свій програмний пакет[39].
Дані Hi-C часто використовуються в аналізі рівнів повногеномної організації хроматину. В результаті обробки існуючими алгоритмами виділяються ТАДи, протяжні лінійні ділянки геному, які пов'язані просторово.[6][48][58]
Hi-C та його похідні постійно удосконалюються. Fit-Hi-C[3] — це метод, заснований на принципі дискретного біннінга даних. Можливі його модифікації з урахуванням відстані взаємодії (уточнення початкового сплайна або spline-1) і уточненням нульовий моделі (spline-2). Результатом Fit-Hi-C є список попарних інтрахромосомних взаємодій з відповідними значеннями p-value і q-value.[59]
3D організація генома може бути встановлена з використанням методів спектрального розкладання матриці контактів. Кожен власний вектор відповідає набору локусів з загальними структурними властивостями (ці локуси необов'язково повинні бути розташовані лінійно один за одним).[60]
Одним з факторів похибок для технології 3C є часті неспецифічні взаємодії між локусами, що з'являються в результаті випадкової поведінки полімеру. Специфічність взаємодії між двома локусами обов'язково повинна бути підтверджена на відповідному рівні статистичної значущості[3].
Нормалізація карти контактів Hi-C
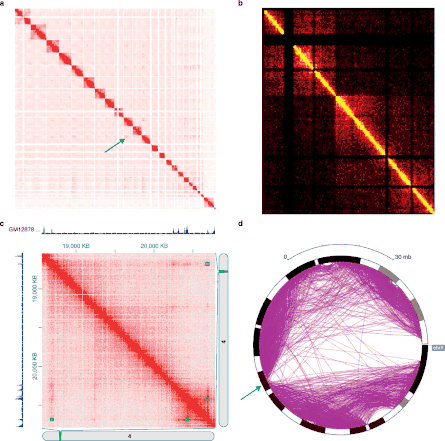
Існує два основних шляхи нормалізації первинних даних теплової карти контактів Hi-C. Перший — припущення стосовно рівної доступності, що означає однакові шанси для кожної позиції в хромосомі брати участь у взаємодії. ВІдповідно, істинний сигнал карти контактів Hi-C повинен являти собою врівноважену матрицю (врівноваженою матрицею вважається така, для якої суми значень по рядках і стовпцях рівні). Прикладом такого алгоритму є алгоритм Сінхорна-Кноппа, який призводить попередню карту контактів до виду врівноваженої матриці.[61]
Інший спосіб використовує припущення про те, що з кожною хромосомною позицією пов'язана деяка зміщеність. Значення карти контактів для кожної координати буде дорівнювати істинному сигналу для даної позиції, помноженому на зміщення для двох сусідніх позицій. До алгоритмів, що використовують модель зі зміщенням, належить алгоритм ітеративной корекції. У процесі його виконання зміщеність по рядках і стовпчикам ітеративно виключається з первинної карти контактів.[60]
Аналіз мотивів ДНК
ДНК-мотиви — це специфічні короткі послідовності ДНК, часто 8-20 нуклеотидів довжиною[62], які статистично завищені у наборі послідовностей із загальною біологічною функцією. В даний час регуляторні мотиви дальньої взаємодії хроматину не вивчені широко. У кількох дослідженнях було зосереджено увагу на з'ясуванні впливу мотивів ДНК у взаємодії промотор-енхансер.
Бейлі та ін. виявили, що мотив ZNF143 в промоторних областях забезпечує специфічність послідовності взаємодій промотор-енхансер.[63] Мутація мотиву ZNF143 зменшила частоту промотор-енхансерних взаємодій, що дозволяє припустити, що ZNF143 є новим фактором циклізації хроматину.
Для аналізу мотивів геномного масштабу у 2016 році Вонг та ін. повідомили про перелік 19 491 пар мотивів ДНК для клітинної лінії K562 щодо взаємодій промотор-енхансер.[64] Як результат, вони запропонували, що кратність зв'язування мотивів (кількість мотивів, сполучених із заданим мотивом) пов'язана з відстанню взаємодії та типом регуляторної області. У наступному році Вонг опублікував ще одну статтю, в якій повідомив про 18 879 пар мотивів у 6 людських клітинних ліній.[65] Новим внеском цієї роботи є MotifHyades — інструмент виявлення мотивів, який можна безпосередньо застосувати до парних послідовностей.
Аналіз ракових геномів
Техніки, засновані на методах 3C, можуть пролити світло на хромосомні перебудови в ракових геномах.[37] Більш того, вони здатні показувати зміни в просторовій близькості регуляторних елементів і їх генів-мішеней, дозволяючи поглибити розуміння структурно-функціональної організації генома в цілому.[66]
Примітки
- de Wit, E.; de Laat, W. (1 січня 2012). A decade of 3C technologies: insights into nuclear organization. Genes & Development 26 (1). с. 11–24. ISSN 0890-9369. doi:10.1101/gad.179804.111. Процитовано 13 січня 2020.
- Hakim, Ofir; Misteli, Tom (2012-03). SnapShot: Chromosome Conformation Capture. Cell (англ.) 148 (5). с. 1068–1068.e2. doi:10.1016/j.cell.2012.02.019. Процитовано 13 січня 2020.
- Ay, Ferhat; Bailey, Timothy L.; Noble, William Stafford (2014-06). Statistical confidence estimation for Hi-C data reveals regulatory chromatin contacts. Genome Research 24 (6). с. 999–1011. ISSN 1549-5469. PMC 4032863. PMID 24501021. doi:10.1101/gr.160374.113. Процитовано 13 січня 2020.
- Dekker, Job (2006-01). The three 'C' s of chromosome conformation capture: controls, controls, controls. Nature Methods (англ.) 3 (1). с. 17–21. ISSN 1548-7091. doi:10.1038/nmeth823. Процитовано 13 січня 2020.
- Dekker, Job (2003-06). A closer look at long-range chromosomal interactions. Trends in Biochemical Sciences (англ.) 28 (6). с. 277–280. doi:10.1016/S0968-0004(03)00089-6. Процитовано 13 січня 2020.
- Rao, Suhas S. P.; Huntley, Miriam H.; Durand, Neva C.; Stamenova, Elena K.; Bochkov, Ivan D.; Robinson, James T.; Sanborn, Adrian L.; Machol, Ido та ін. (18 грудня 2014). A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell 159 (7). с. 1665–1680. ISSN 1097-4172. PMC 5635824. PMID 25497547. doi:10.1016/j.cell.2014.11.021. Процитовано 13 січня 2020.
- Varoquaux, Nelle; Ay, Ferhat; Noble, William Stafford; Vert, Jean-Philippe (15 червня 2014). A statistical approach for inferring the 3D structure of the genome. Bioinformatics (Oxford, England) 30 (12). с. i26–33. ISSN 1367-4811. PMC 4229903. PMID 24931992. doi:10.1093/bioinformatics/btu268. Процитовано 13 січня 2020.
- Davies, James O J; Oudelaar, A Marieke; Higgs, Douglas R; Hughes, Jim R (2017-02). How best to identify chromosomal interactions: a comparison of approaches. Nature Methods (англ.) 14 (2). с. 125–134. ISSN 1548-7091. doi:10.1038/nmeth.4146. Процитовано 13 січня 2020.
- Denker, Annette; de Laat, Wouter (06 15, 2016). The second decade of 3C technologies: detailed insights into nuclear organization. Genes & Development 30 (12). с. 1357–1382. ISSN 1549-5477. PMC 4926860. PMID 27340173. doi:10.1101/gad.281964.116. Процитовано 13 січня 2020.
- WHIPPLE, ROBERT S. (1933-02). The History of the Microscope: Compiled from Original Instruments and Documents, up to the Introduction of the Achromatic Microscope. Nature 131 (3303). с. 219–221. ISSN 0028-0836. doi:10.1038/131219a0. Процитовано 13 січня 2020.
- Flemming, Walther (1871). Ueber Bindesubstanzen und Gefässwandung bei Mollusken.. Rostock :: Carl Boldis Buchdr.,.
- Martins, L.A.-C.P. (1999-06). Did Sutton and Boveri propose the so-called Sutton-Boveri chromosome hypothesis?. Genetics and Molecular Biology 22 (2). с. 261–272. ISSN 1415-4757. doi:10.1590/S1415-47571999000200022. Процитовано 13 січня 2020.
- Broccias, Cristiano (17 лютого 2012). Nikolas Gisborne, The event structure of perception verbs. Oxford: Oxford University Press, 2010. Pp. x + 317.. English Language and Linguistics 16 (1). с. 176–183. ISSN 1360-6743. doi:10.1017/s1360674311000347. Процитовано 13 січня 2020.
- Eissenberg, Joel C; Elgin, Sarah (19 квітня 2001). Heterochromatin and Euchromatin. Encyclopedia of Life Sciences (John Wiley & Sons, Ltd). ISBN 0-470-01590-X. Процитовано 13 січня 2020.
- Deichmann, Ute (08 01, 2016). Epigenetics: The origins and evolution of a fashionable topic. Developmental Biology 416 (1). с. 249–254. ISSN 1095-564X. PMID 27291929. doi:10.1016/j.ydbio.2016.06.005. Процитовано 13 січня 2020.
- Lu, Haoyang; Liu, Xinzhou; Deng, Yulin; Qing, Hong (5 грудня 2013). DNA methylation, a hand behind neurodegenerative diseases. Frontiers in Aging Neuroscience 5. с. 85. ISSN 1663-4365. PMC 3851782. PMID 24367332. doi:10.3389/fnagi.2013.00085. Процитовано 13 січня 2020.
- "The Francis Crick Papers: The Discovery of the Double Helix, 1951-1953".
- Cremer, Thomas; Cremer, Marion (2010-03). Chromosome territories. Cold Spring Harbor Perspectives in Biology 2 (3). с. a003889. ISSN 1943-0264. PMC 2829961. PMID 20300217. doi:10.1101/cshperspect.a003889. Процитовано 13 січня 2020.
- Cullen, K. E.; Kladde, M. P.; Seyfred, M. A. (9 липня 1993). Interaction between transcription regulatory regions of prolactin chromatin. Science (New York, N.Y.) 261 (5118). с. 203–206. ISSN 0036-8075. PMID 8327891. doi:10.1126/science.8327891. Процитовано 13 січня 2020.
- Dekker, Job; Rippe, Karsten; Dekker, Martijn; Kleckner, Nancy (15 лютого 2002). Capturing chromosome conformation. Science (New York, N.Y.) 295 (5558). с. 1306–1311. ISSN 1095-9203. PMID 11847345. doi:10.1126/science.1067799. Процитовано 13 січня 2020.
- Osborne, Cameron S.; Ewels, Philip A.; Young, Alice N. C. (2011-01). Meet the neighbours: tools to dissect nuclear structure and function. Briefings in Functional Genomics 10 (1). с. 11–17. ISSN 2041-2657. PMC 3080762. PMID 21258046. doi:10.1093/bfgp/elq034. Процитовано 13 січня 2020.
- Simonis, Marieke; Klous, Petra; Splinter, Erik; Moshkin, Yuri; Willemsen, Rob; de Wit, Elzo; van Steensel, Bas; de Laat, Wouter (2006-11). Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics 38 (11). с. 1348–1354. ISSN 1061-4036. PMID 17033623. doi:10.1038/ng1896. Процитовано 13 січня 2020.
- Dostie, Josée; Richmond, Todd A.; Arnaout, Ramy A.; Selzer, Rebecca R.; Lee, William L.; Honan, Tracey A.; Rubio, Eric D.; Krumm, Anton та ін. (2006-10). Chromosome Conformation Capture Carbon Copy (5C): a massively parallel solution for mapping interactions between genomic elements. Genome Research 16 (10). с. 1299–1309. ISSN 1088-9051. PMC 1581439. PMID 16954542. doi:10.1101/gr.5571506. Процитовано 13 січня 2020.
- Albert, Istvan; Mavrich, Travis N.; Tomsho, Lynn P.; Qi, Ji; Zanton, Sara J.; Schuster, Stephan C.; Pugh, B. Franklin (29 березня 2007). Translational and rotational settings of H2A.Z nucleosomes across the Saccharomyces cerevisiae genome. Nature 446 (7135). с. 572–576. ISSN 1476-4687. PMID 17392789. doi:10.1038/nature05632. Процитовано 13 січня 2020.
- Lieberman-Aiden, Erez; van Berkum, Nynke L.; Williams, Louise; Imakaev, Maxim; Ragoczy, Tobias; Telling, Agnes; Amit, Ido; Lajoie, Bryan R. та ін. (9 жовтня 2009). Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science (New York, N.Y.) 326 (5950). с. 289–293. ISSN 1095-9203. PMC 2858594. PMID 19815776. doi:10.1126/science.1181369. Процитовано 13 січня 2020.
- Fullwood, Melissa J.; Liu, Mei Hui; Pan, You Fu; Liu, Jun; Xu, Han; Mohamed, Yusoff Bin; Orlov, Yuriy L.; Velkov, Stoyan та ін. (5 листопада 2009). An oestrogen-receptor-alpha-bound human chromatin interactome. Nature 462 (7269). с. 58–64. ISSN 1476-4687. PMC 2774924. PMID 19890323. doi:10.1038/nature08497. Процитовано 13 січня 2020.
- Dixon, Jesse R.; Selvaraj, Siddarth; Yue, Feng; Kim, Audrey; Li, Yan; Shen, Yin; Hu, Ming; Liu, Jun S. та ін. (11 квітня 2012). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature 485 (7398). с. 376–380. ISSN 1476-4687. PMC 3356448. PMID 22495300. doi:10.1038/nature11082. Процитовано 13 січня 2020.
- Nora, Elphège P.; Lajoie, Bryan R.; Schulz, Edda G.; Giorgetti, Luca; Okamoto, Ikuhiro; Servant, Nicolas; Piolot, Tristan; van Berkum, Nynke L. та ін. (11 квітня 2012). Spatial partitioning of the regulatory landscape of the X-inactivation centre. Nature 485 (7398). с. 381–385. ISSN 1476-4687. PMC 3555144. PMID 22495304. doi:10.1038/nature11049. Процитовано 13 січня 2020.
- Nagano, Takashi; Lubling, Yaniv; Stevens, Tim J.; Schoenfelder, Stefan; Yaffe, Eitan; Dean, Wendy; Laue, Ernest D.; Tanay, Amos та ін. (3 жовтня 2013). Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature 502 (7469). с. 59–64. ISSN 1476-4687. PMC 3869051. PMID 24067610. doi:10.1038/nature12593. Процитовано 13 січня 2020.
- Gavrilov, Alexey; Eivazova, Elvira; Pirozhkova, Iryna; Lipinski, Marc; Razin, Sergey; Vassetzky, Yegor (2009). У Collas, Philippe. Chromosome Conformation Capture (from 3C to 5C) and Its ChIP-Based Modification. Chromatin Immunoprecipitation Assays 567. Totowa, NJ: Humana Press. с. 171–188. ISBN 978-1-60327-413-5. doi:10.1007/978-1-60327-414-2_12.
- Naumova, Natalia; Smith, Emily M.; Zhan, Ye; Dekker, Job (2012-11). Analysis of long-range chromatin interactions using Chromosome Conformation Capture. Methods (англ.) 58 (3). с. 192–203. doi:10.1016/j.ymeth.2012.07.022. Процитовано 13 січня 2020.
- Belton, Jon-Matthew; Dekker, Job (2015-06). Chromosome Conformation Capture (3C) in Budding Yeast. Cold Spring Harbor Protocols (англ.) 2015 (6). с. pdb.prot085175. ISSN 1940-3402. doi:10.1101/pdb.prot085175. Процитовано 13 січня 2020.
- Gavrilov, Alexey A.; Golov, Arkadiy K.; Razin, Sergey V. (26 березня 2013). Actual Ligation Frequencies in the Chromosome Conformation Capture Procedure. У Dean, Ann. PLoS ONE (англ.) 8 (3). с. e60403. ISSN 1932-6203. doi:10.1371/journal.pone.0060403. Процитовано 13 січня 2020.
- Stadhouders, Ralph; Kolovos, Petros; Brouwer, Rutger; Zuin, Jessica; van den Heuvel, Anita; Kockx, Christel; Palstra, Robert-Jan; Wendt, Kerstin S та ін. (2013-03). Multiplexed chromosome conformation capture sequencing for rapid genome-scale high-resolution detection of long-range chromatin interactions. Nature Protocols (англ.) 8 (3). с. 509–524. ISSN 1754-2189. doi:10.1038/nprot.2013.018. Процитовано 13 січня 2020.
- Zhao, Zhihu; Tavoosidana, Gholamreza; Sjölinder, Mikael; Göndör, Anita; Mariano, Piero; Wang, Sha; Kanduri, Chandrasekhar; Lezcano, Magda та ін. (2006-11). Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nature Genetics (англ.) 38 (11). с. 1341–1347. ISSN 1061-4036. doi:10.1038/ng1891. Процитовано 13 січня 2020.
- van de Werken, Harmen J G; Landan, Gilad; Holwerda, Sjoerd J B; Hoichman, Michael; Klous, Petra; Chachik, Ran; Splinter, Erik; Valdes-Quezada, Christian та ін. (2012-10). Robust 4C-seq data analysis to screen for regulatory DNA interactions. Nature Methods (англ.) 9 (10). с. 969–972. ISSN 1548-7091. doi:10.1038/nmeth.2173. Процитовано 13 січня 2020.
- Harewood, Louise; Kishore, Kamal; Eldridge, Matthew D.; Wingett, Steven; Pearson, Danita; Schoenfelder, Stefan; Collins, V. Peter; Fraser, Peter (2017-12). Hi-C as a tool for precise detection and characterisation of chromosomal rearrangements and copy number variation in human tumours. Genome Biology (англ.) 18 (1). с. 125. ISSN 1474-760X. doi:10.1186/s13059-017-1253-8. Процитовано 13 січня 2020.
- Burton, Joshua N; Adey, Andrew; Patwardhan, Rupali P; Qiu, Ruolan; Kitzman, Jacob O; Shendure, Jay (2013-12). Chromosome-scale scaffolding of de novo genome assemblies based on chromatin interactions. Nature Biotechnology (англ.) 31 (12). с. 1119–1125. ISSN 1087-0156. doi:10.1038/nbt.2727. Процитовано 13 січня 2020.
- Schmitt, Anthony D.; Hu, Ming; Ren, Bing (2016-12). Genome-wide mapping and analysis of chromosome architecture. Nature Reviews Molecular Cell Biology (англ.) 17 (12). с. 743–755. ISSN 1471-0072. doi:10.1038/nrm.2016.104. Процитовано 13 січня 2020.
- Hughes, Jim R; Roberts, Nigel; McGowan, Simon; Hay, Deborah; Giannoulatou, Eleni; Lynch, Magnus; De Gobbi, Marco; Taylor, Stephen та ін. (2014-02). Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics (англ.) 46 (2). с. 205–212. ISSN 1061-4036. doi:10.1038/ng.2871. Процитовано 13 січня 2020.
- Davies, James O J; Telenius, Jelena M; McGowan, Simon J; Roberts, Nigel A; Taylor, Stephen; Higgs, Douglas R; Hughes, Jim R (2016-01). Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods (англ.) 13 (1). с. 74–80. ISSN 1548-7091. doi:10.1038/nmeth.3664. Процитовано 13 січня 2020.
- the GIANT Consortium (2009-01). Six new loci associated with body mass index highlight a neuronal influence on body weight regulation. Nature Genetics (англ.) 41 (1). с. 25–34. ISSN 1061-4036. doi:10.1038/ng.287. Процитовано 13 січня 2020.
- Jäger, Roland; Migliorini, Gabriele; Henrion, Marc; Kandaswamy, Radhika; Speedy, Helen E.; Heindl, Andreas; Whiffin, Nicola; Carnicer, Maria J. та ін. (2015-05). Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications (англ.) 6 (1). с. 6178. ISSN 2041-1723. doi:10.1038/ncomms7178. Процитовано 13 січня 2020.
- Tiwari, V. K.; Baylin, S. B. (1 березня 2009). Combined 3C-ChIP-Cloning (6C) Assay: A Tool to Unravel Protein-Mediated Genome Architecture. Cold Spring Harbor Protocols (англ.) 2009 (3). с. pdb.prot5168–pdb.prot5168. ISSN 1559-6095. doi:10.1101/pdb.prot5168. Процитовано 13 січня 2020.
- Horike, Shin-ichi; Cai, Shutao; Miyano, Masaru; Cheng, Jan-Fang; Kohwi-Shigematsu, Terumi (2005-01). Loss of silent-chromatin looping and impaired imprinting of DLX5 in Rett syndrome. Nature Genetics (англ.) 37 (1). с. 31–40. ISSN 1061-4036. doi:10.1038/ng1491. Процитовано 13 січня 2020.
- Mumbach, Maxwell R.; Rubin, Adam J.; Flynn, Ryan A.; Dai, Chao; Khavari, Paul A.; Greenleaf, William J.; Chang, Howard Y. (2016-11). HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods 13 (11). с. 919–922. ISSN 1548-7105. PMC 5501173. PMID 27643841. doi:10.1038/nmeth.3999. Процитовано 13 січня 2020.
- Tolhuis, Bas; Palstra, Robert-Jan; Splinter, Erik; Grosveld, Frank; de Laat, Wouter (2002-12). Looping and Interaction between Hypersensitive Sites in the Active β-globin Locus. Molecular Cell (англ.) 10 (6). с. 1453–1465. doi:10.1016/S1097-2765(02)00781-5. Процитовано 13 січня 2020.
- Cavalli, Giacomo; Misteli, Tom (2013-03). Functional implications of genome topology. Nature Structural & Molecular Biology (англ.) 20 (3). с. 290–299. ISSN 1545-9993. doi:10.1038/nsmb.2474. Процитовано 13 січня 2020.
- Dekker, Job; Marti-Renom, Marc A.; Mirny, Leonid A. (2013-06). Exploring the three-dimensional organization of genomes: interpreting chromatin interaction data. Nature Reviews Genetics (англ.) 14 (6). с. 390–403. ISSN 1471-0056. doi:10.1038/nrg3454. Процитовано 13 січня 2020.
- Guo, Ya; Xu, Quan; Canzio, Daniele; Shou, Jia; Li, Jinhuan; Gorkin, David U.; Jung, Inkyung; Wu, Haiyang та ін. (2015-08). CRISPR Inversion of CTCF Sites Alters Genome Topology and Enhancer/Promoter Function. Cell (англ.) 162 (4). с. 900–910. doi:10.1016/j.cell.2015.07.038. Процитовано 13 січня 2020.
- Krijger, Peter Hugo Lodewijk; de Laat, Wouter (2016-12). Regulation of disease-associated gene expression in the 3D genome. Nature Reviews Molecular Cell Biology (англ.) 17 (12). с. 771–782. ISSN 1471-0072. doi:10.1038/nrm.2016.138. Процитовано 13 січня 2020.
- Fritsch, E. F.; Lawn, R. M.; Maniatis, T. (14 червня 1979). Characterisation of deletions which affect the expression of fetal globin genes in man. Nature 279 (5714). с. 598–603. ISSN 0028-0836. PMID 450109. doi:10.1038/279598a0. Процитовано 13 січня 2020.
- Van der Ploeg, L. H.; Konings, A.; Oort, M.; Roos, D.; Bernini, L.; Flavell, R. A. (14 лютого 1980). gamma-beta-Thalassaemia studies showing that deletion of the gamma- and delta-genes influences beta-globin gene expression in man. Nature 283 (5748). с. 637–642. ISSN 0028-0836. PMID 6153459. doi:10.1038/283637a0. Процитовано 13 січня 2020.
- Jeong, Y. (15 лютого 2006). A functional screen for sonic hedgehog regulatory elements across a 1 Mb interval identifies long-range ventral forebrain enhancers. Development (англ.) 133 (4). с. 761–772. ISSN 0950-1991. doi:10.1242/dev.02239. Процитовано 13 січня 2020.
- Wieczorek, Dagmar; Pawlik, Barbara; Li, Yun; Akarsu, Nurten A.; Caliebe, Almuth; May, Klaus J.W.; Schweiger, Bernd; Vargas, Fernando R. та ін. (2010-01). A specific mutation in the distant sonic hedgehog ( SHH ) cis -regulator (ZRS) causes Werner mesomelic syndrome (WMS) while complete ZRS duplications underlie Haas type polysyndactyly and preaxial polydactyly (PPD) with or without triphalangeal thumb. Human Mutation (англ.) 31 (1). с. 81–89. doi:10.1002/humu.21142. Процитовано 13 січня 2020.
- Zhang, Xiaoyang; Choi, Peter S; Francis, Joshua M; Imielinski, Marcin; Watanabe, Hideo; Cherniack, Andrew D; Meyerson, Matthew (2016-02). Identification of focally amplified lineage-specific super-enhancers in human epithelial cancers. Nature Genetics (англ.) 48 (2). с. 176–182. ISSN 1061-4036. PMC PMC4857881. PMID 26656844. doi:10.1038/ng.3470. Процитовано 13 січня 2020.
- Mansour, M. R.; Abraham, B. J.; Anders, L.; Berezovskaya, A.; Gutierrez, A.; Durbin, A. D.; Etchin, J.; Lawton, L. та ін. (12 грудня 2014). An oncogenic super-enhancer formed through somatic mutation of a noncoding intergenic element. Science (англ.) 346 (6215). с. 1373–1377. ISSN 0036-8075. PMC PMC4720521. PMID 25394790. doi:10.1126/science.1259037. Процитовано 13 січня 2020.
- Dixon, Jesse R.; Selvaraj, Siddarth; Yue, Feng; Kim, Audrey; Li, Yan; Shen, Yin; Hu, Ming; Liu, Jun S. та ін. (2012-05). Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature (англ.) 485 (7398). с. 376–380. ISSN 0028-0836. doi:10.1038/nature11082. Процитовано 13 січня 2020.
- Yardımcı, Galip Gürkan; Noble, William Stafford (2017-12). Software tools for visualizing Hi-C data. Genome Biology (англ.) 18 (1). с. 26. ISSN 1474-760X. doi:10.1186/s13059-017-1161-y. Процитовано 13 січня 2020.
- Imakaev, Maxim; Fudenberg, Geoffrey; McCord, Rachel Patton; Naumova, Natalia; Goloborodko, Anton; Lajoie, Bryan R; Dekker, Job; Mirny, Leonid A (2012-10). Iterative correction of Hi-C data reveals hallmarks of chromosome organization. Nature Methods (англ.) 9 (10). с. 999–1003. ISSN 1548-7091. doi:10.1038/nmeth.2148. Процитовано 13 січня 2020.
- Lajoie, Bryan R.; Dekker, Job; Kaplan, Noam (2015-01). The Hitchhiker’s guide to Hi-C analysis: Practical guidelines. Methods (англ.) 72. с. 65–75. doi:10.1016/j.ymeth.2014.10.031. Процитовано 13 січня 2020.
- Zambelli, Federico; Pesole, Graziano; Pavesi, Giulio (2013-03). Motif discovery and transcription factor binding sites before and after the next-generation sequencing era. Briefings in Bioinformatics 14 (2). с. 225–237. ISSN 1477-4054. PMC 3603212. PMID 22517426. doi:10.1093/bib/bbs016. Процитовано 13 січня 2020.
- Bailey, Swneke D.; Zhang, Xiaoyang; Desai, Kinjal; Aid, Malika; Corradin, Olivia; Cowper-Sal·lari, Richard; Akhtar-Zaidi, Batool; Scacheri, Peter C. та ін. (3 лютого 2015). ZNF143 provides sequence specificity to secure chromatin interactions at gene promoters. Nature Communications (англ.) 6 (1). с. 1–10. ISSN 2041-1723. PMC PMC4431651. PMID 25645053. doi:10.1038/ncomms7186. Процитовано 13 січня 2020.
- Wong, Ka-Chun; Li, Yue; Peng, Chengbin (26 вересня 2015). Identification of coupling DNA motif pairs on long-range chromatin interactions in human K562 cells. Bioinformatics 32 (3). с. 321–324. ISSN 1367-4803. doi:10.1093/bioinformatics/btv555. Процитовано 13 січня 2020.
- Wong, Ka-Chun (1 жовтня 2017). MotifHyades: expectation maximization for de novo DNA motif pair discovery on paired sequences. Bioinformatics (англ.) 33 (19). с. 3028–3035. ISSN 1367-4803. doi:10.1093/bioinformatics/btx381. Процитовано 13 січня 2020.
- Taberlay, Phillippa C.; Achinger-Kawecka, Joanna; Lun, Aaron T.L.; Buske, Fabian A.; Sabir, Kenneth; Gould, Cathryn M.; Zotenko, Elena; Bert, Saul A. та ін. (2016-06). Three-dimensional disorganization of the cancer genome occurs coincident with long-range genetic and epigenetic alterations. Genome Research (англ.) 26 (6). с. 719–731. ISSN 1088-9051. doi:10.1101/gr.201517.115. Процитовано 13 січня 2020.