НАДН-дегідрогеназний комплекс
НАДH-дегідрогеназний комплекс, відомий також як комплекс I чи НАДH-убіхінон-оксидоредуктаза — перший багатобілковий комплекс дихального ланцюга мітохондрій[1]. Багато копій комплексу містяться в мембранах прокаріотичних організмів, здатних до кисневого дихання та внутрішніх мембранах мітохондрій еукаріотичних клітин[2]. Коли йдеться про комплекс як про біохімічну структуру людини, комплекс I часто називають НАДH-дегідрогеназою.
 Сірим кольором показана внутрішня мембрана мітохондрії. Згори — мітохондріальний матрикс, знизу — міжмембранний простір. | |
| Ідентифікатори | |
|---|---|
| Код КФ | 1.6.5.3 |
| Бази ферментів | |
| IntEnz | IntEnz view |
| BRENDA | BRENDA entry |
| ExPASy | NiceZyme view |
| MetaCyc | metabolic pathway |
| KEGG | KEGG entry |
| PRIAM | profile |
| PDB structures | RCSB PDB PDBe PDBj PDBsum |
| Gene Ontology | AmiGO • EGO |
| Пошук | |
| PMC | статті |
| PubMed | статті |
| NCBI | NCBI proteins |
Цей комплекс відіграє центральну роль у процесах клітинного дихання та окисного фосфорилювання, адже майже 40 % протонного градієнту для синтезу АТФ утворюється саме цим комплексом[3]. Комплекс I окиснює НАДH і відновлює одну молекулу убіхінону, яка вивільнюється в мембрану. На кожну окиснену молекулу НАДH комплекс переносить через мембрану чотири протони.
Комплекс I виділили з низки об'єктів: мітохондрій серця бика, цукрового буряка (Beta vulgaris), картоплі (Solanum tuberosum), бобів (Vicia faba), різушки Таля (Arabidopsis thaliana) та рису (Oryza sativa), а також із мітохондрій гриба нейроспори (Neurospora crassa) та мембран кишкової палички (Escherichia coli)[4].
Структурна організація
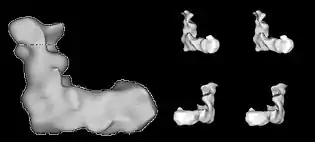
У прокаріотів комплекс I складається з 14 основних субодиниць, що утворюють ядро комплексу, без якого він не в змозі функціонувати. Сім субодиниць є надзвичайно гідрофобними та локалізуються в мембрані, а сім відносно гідрофільних перебувають поза мембраною. В еукаріотів унаслідок еволюції комплекс покрився «шубою» з близько 30 допоміжних субодиниць, їх кількість може змінюватись залежно від об'єкту. Так, у ссавців цей фермент складається з 44 субодиниць, а в гриба Yarrowia lipolytica — з 48[5]. Унаслідок цієї «надбудови» молекулярна маса комплексу I збільшилась майже удвічі: з ~550 кДа в бактерій до ~1 МДа в мітохондріях[6].
Електронна мікроскопія довела, що комплекс I (як із бактерій, так і з мітохондрій) має характерну L-подібну форму. Через цю форму, а також через ніби пом'яту молекулярну поверхню, комплекс I одержав від вчених назву «старий черевик». Гідрофобна «підошва» представлена білками, вмонтованими в мембрану, а гідрофільна частина — «кісточка» — обернена до матриксу[4].
Чотири субодиниці убіхінон-зв'язувального модуля разом із субодиницями мембранної частини ферменту утворюють сайт зв'язування убіхінону, де він взаємодіє із залізосірковим кластером N2, приймає два електрони та відновлюється до убіхінолу. Кластер N2, останній у ряді кластерів, по яких електрони від НАДH переходять до убіхінону, припіднесений над мембраною на ~15 Å. Сама порожнина, в якій зв'язується убіхінон, є завдовжки 30 Å і може вмістити всю молекулу разом із довгим гідрофобним хвостом із семи ізопренових одиниць. Порожнина має вузький вхід, тож довгий гідрофобний ланцюг повинен набути певної конформації, яка збережеться протягом усієї ферментативної реакції. Наявність такого довгого та вузького сайту зв'язування є характерною рисою комплексу I. Усередині порожнини убіхінон взаємодіє з консервативними залишками тирозину та гістидину[7].
У грибів, тварин і судинних рослин принаймні 7 із 44 субодиниць, які утворюють мембранний домен, кодуються мітохондріальним геномом[8]. У ссавців цих субодиниць є рівно сім[9][10]. У рослин ДНК мітохондрій кодує дев'ять субодиниць: окрім семи субодиниць, які входять до складу гідрофобної частини комплексу, в ній закодовані дві субодиниці, гомологічні субодиницям 49 кДа та 30 кДа у ссавців, а решта компонентів перебувають під контролем ядерних генів[4]. Однак ці дані, отриманні при дослідженні пластомів картоплі та різушки Таля, можуть виявитись недостовірними для інших видів рослин, а кількість закодованих у мітохондріях субодиниць може змінюватись від виду до виду. Так, у печіночника Marchantia polymorpha субодиниця NAD7, гомологічна поліпептиду 49 кДа, кодується ядерним геномом і транспортується в мітохондрії, а відповідний ген мітохондріальної ДНК перетворився в псевдоген і є нефункціональним[11].
Дослідження виявили, що за своїми властивостями комплекс I суттєво не відрізняється в об'єктів тваринного та рослинного походження[4]. Однак у рослин наявні деякі специфічні субодиниці, які в деяких випадках надають комплексу функціональних особливостей. Аналіз комплексу I в Arabidopsis показує, що понад 30 % субодиниць є специфічними для рослин[12]. Наприклад, частиною мембранного модуля рослинного комплексу I є так званий γ-карбоангідразний структурний модуль і L-галактоно-1,4-лактондегідрогеназа, яка водночас являє собою останній фермент мітохондріального шляху біосинтезу аскорбінової кислоти[13].
Таблиця основних (корових) субодиниць
| Основні субодиниці[6][7][14] | ||||||
|---|---|---|---|---|---|---|
| Корова | Людина | Кишкова паличка | Різушка Таля | Опис | Кофактори | |
| Периферична рука | ||||||
| Дегідрогеназний модуль | 75 кДа | NDUFS1 | NuoG | 75 кДа | транспорт електронів | Fe2S2 (N1b), 2 x Fe4S4 (N4 и N5), (N7) |
| 51 kDa | NDUFV1 | NuoF | 51 кДа | зв'язування НАДН, транспорт електронів | флавін, Fe4S4 (N3) | |
| 24 kDa | NDUFV2 | NuoE | 24 кДа | Несе допоміжний кластер N1a | Fe2S2 (N1a) | |
| Убіхінон-зв'язувальний модуль | 49 кДа | NDUFS2 | NuoD (NuoCD) | NAD7 | ||
| 30 kDa | NDUFS3 | NuoC | NAD9 | |||
| TYKY | NDUFS8 | NuoI | 28,5 кДа | транспорт електронів | 2 x Fe4S4 (N6a и N6b) | |
| PSST | NDUFS7 | NuoB | 20 кДа | транспорт електронів | Fe4S4 (N2) | |
| Мембранна рука | ||||||
| - | ND1 | MT-ND1 | NuoH | NAD1 | ||
| Протон-транслокувальний модуль | ND2 | MT-ND2 | NuoN | NAD2 | антипорт-подібний білок | |
| ND3 | MT-ND3 | NuoA | NAD3 | |||
| ND4 | MT-ND4 | NuoM | NAD4 | антипорт-подібний білок | ||
| ND4L | MT-ND4L | NuoK | NAD4L | |||
| ND5 | MT-ND5 | NuoL | NAD5 | антипорт-подібний білок | ||
| ND6 | MT-ND6 | NuoJ | NAD6 | |||
Таблиця допоміжних субодиниць
Усі мітохондріальні комплекси I мають багато допоміжних субодиниць, які не є необхідними для каталітичної активності та відрізняються в різних видів. Очевидно, що вони піддаються певному функціональному навантаженню, оскільки їхні мутації призводять до спадкових захворювань. Для деяких субодиниць виявлено наявність певних функцій, так B16.6 (GRIM-19) бере участь в апоптозі, а субодиниця 39 кДа (NDUFA9) — у регуляції активності комплексу[15]. Щодо інших субодиниць, то сьогодні активно обговорюється їх можлива роль у регуляції, збірці, стабілізації та захисті від активних форм кисню. Варто зазначити, що допоміжні субодиниці значно збільшують енергетичні витрати клітини на синтез, складання та деградацію комплексу. Такі затрати, тим не менш, можуть окупитись у випадку ядерної клітини, в якій процес синтезу білка добре контролюється. З іншого боку, якщо допоміжні субодиниці є необхідними для стабілізації комплексу I, залишається незрозумілим, яким чином бактеріальні комплекси, які складаються з мінімально потрібної кількості поліпептидів, успішно функціонують без них. На сьогодні вчені не дають чіткої відповіді на ці запитання[7].
| Допоміжні субодиниці[6][7][14][16] | ||||
|---|---|---|---|---|
| No. | Корова | Білок людини | Опис | Родина білків Pfam |
| Субодиниці | ||||
| 15 | 13A | NDUFS6 | бере участь у складанні та стабілізує комплекс | PF10276 |
| 16 | B17.2 | NDUFA12 | PF05071 | |
| 17 | AQDQ | NDUFS4 | можливо, фосфорилюється протеїнкіназою А (ПКА), стабілізує комплекс під час складання | PF04800 |
| 18 | 39kDa | NDUFA9 | сполучає мембранний та периферичний домени, бере участь у активації/деактивації комплексу | PF01370 |
| 19 | ACPM | ACPM | ацил-переносний білок | PF00550 |
| 20 | B8 | NDUFA2 | PF05047 | |
| 21 | MFWE | NDUFA1 | цАМФ залежне фосфорилювання ПКА | PF15879 |
| 22 | B12 | NDUFB3 | PF08122 | |
| 23 | AB13 | NDUFA5 | бере участь у активації/деактивації комплексу | PF04716 |
| 24 | B14 | NDUFA6 | PF05347 | |
| 25 | B14.7 | NDUFA11 | PF02466 | |
| 26 | ESSS | NDUFB11 | цАМФ залежне фосфорилювання протеїнкінази A | PF10183 |
| 27 | PFFD | NDUFS5 | PF10200 | |
| 28 | B15 | NDUFB4 | PF07225 | |
| 29 | B16.6 | NDUFA13 | бере участь в апоптозі | PF06212 |
| 30 | B18 | NDUFB7 | PF05676 | |
| 31 | PGIV | NDUFA8 | PF06747 | |
| 32 | B22 | NDUFB9 | PF05347 | |
| 33 | PDSW | NDUFB10 | фосфорилюється Src у ракових клітинах | PF10249 |
| 34 | ASHI | NDUFB8 | PF05821 | |
| 35 | B14.5B | NDUFC2 | у нормі фосфорильована | PF06374 |
| 36 | AGGG | NDUFB2 | PF14813 | |
| 37 | B14.5A | NDUFA7 | у нормі фосфорильована | PF07347 |
| 38 | B9 | NDUFA3 | PF14987 | |
| 39 | MLRQ | NDUFA4 | PF06522 | |
| 40 | SGDH | NDUFB5 | PF09781 | |
| 41 | MNLL | NDUFB1 | PF08040 | |
| 42 | KFYI | NDUFC1 | у нормі фосфорильована | PF15088 |
| 43 | 42kD | NDUFA10 | PF01712 | |
| 44 | NDUFA4L2 | NDUFA4L2 | PF15880 | |
| 45 | NDUFV3 | NDUFV3 | - | |
| 46 | NDUFB6 | NDUFB6 | PF09782 | |
| Допоміжні білки, | ||||
| 47 | NDUFAF1 | CIA30 | фактор складання комплексу 1 | PF08547 |
| 48 | NDUFAF2 | NDUFAF2 | фактор складання комплексу 2 | PF05071 |
| 49 | NDUFAF3 | NDUFAF3 | фактор складання комплексу 3 | PF05071 |
| 50 | NDUFAF4 | NDUFAF4 | фактор складання комплексу 4 | PF06784 |
Деякі допоміжні субодиниці фосфорилюються різними кіназами, що ніколи не відбувається з субодиницями кор-ферменту. Побутують здогадки, що таким чином відбувається регуляція роботи комплексу. У вигляді однієї з субодиниць присутній у комплексі ацил-переносний білок (NDUFAB1) із фосфорильованої пантотенової кислоти як простетичної групи. Припускають, що він бере участь у синтезі ліпоєвої кислоти, репарації пошкоджених ліпідів мембрани або ж модифікує інші білки залишками міристинової кислоти. Варто зазначити, що функціонування цього білка не залежить від прямого фізичного контакту з комплексом I і значна його частина присутня у вільному вигляді серед мітохондріального матриксу[16].
Кофактори

Усі простетичні групи НАДН-дегідрогеназного комплексу (один флавінмононуклеотид і від 8 до 9 залізосіркових кластерів) містяться в периферичному водорозчинному домені. У ссавців, як і у всіх хребетних, їх є вісім[9]. Сім кластерів утворюють електрон-транспортний ланцюг завдовжки ~96 Å від ФМН до місця зв'язування убіхінону. Відштовхуючись від сучасних даних, вважається, що перенесення електрона відбувається по такому шляху: НАДН → ФМН → N3 → N1b → N4 → N5 → N6a → N6b → N2 → Q. Спочатку відбувається передання двох електронів на флавін, а відтак вони один за одним передаються через ланцюжок кластерів до сайту зв'язування хінону та відновлюють його до стану Q−2. Кластер N1a міститься недалеко від флавінового кофактора та не певній відстані від магістрального ланцюга перенесення електронів. Цей кластер є висококонсервативним у різних видів; вважають, що він контролює швидкість транспорту електрона серед комплексу, перекидуючись електроном із ФМН[6]. Існує модель, відповідно до якої один із електронів із флавіну йде магістральним шляхом на хінон, а інший запасається в кластері N1a, а відтак повертається в основний ланцюг через флавосеміхінон. Можливо, такий механізм дає можливість сповільнити утворення активних форм кисню на відновленому флавіні. Тим більше, це сприяє стабілізації (до мілісекунди) стану, за якого кластер N2 відновлений, але немає другого електрона, щоб завершити відновлення убіхінону. Такий стан може бути необіхідним для конформаційних змін, спряжених із транспортом протонів.
Частина кластерів із ланцюга (N3, N4 и N6a) мають високий окисно-відновний потенціал (редокс-потенціал) на рівні —0,25 В, у той час як три інших (N1b, N5 і N6b) мають нижчі потенціали. Унаслідок цього редокс-потенціал на шляху електрона міняється за типом американських гірок. Така крива зміни енергетичного стану є характерною для багатьох окисно-відновних ферментів: вона дає змогу оптимізувати швидкість транспорту електрона та домогтися ефективного перенесення енергії[6].
Кластер N5 має дуже низький потенціал, тому лімітує швидкість загального потоку електронів по всьому ланцюгу. Замість звичайних для залізосіркових центрів лігандів (чотирьох залишків цистеїну), він є скоординований трьома залишками цистеїну та одним залишком гістидину, а також оточений зарядженими полярними залишками, хоч і розташований у глибині ферменту[6].
Незвичайні ліганди має і термінальний кластер ланцюга — N2. Його редокс-потенціал є найвищим серед усіх кластерів (від —0,1 до —0,15 В). Він зв'язаний із чотирма послідовно розміщеними в поліпептидному ланцюгу залишками цистеїну, що створює напружену конформацію. Через це при його відновленні відбуваються конформаційні зміни сусідніх ланцюгів, можливо, пов'язані з транспортом протона[6].
Кластер N7 присутній лише в комплексі I деяких бактерій. Він є значно віддалений від решти кластерів і тому не може обмінюватись із ними електронами, тож скоріш за все є реліктом. У деяких бактеріальних комплексах, споріднених із комплексом I, між N7 і рештою кластерів виявили чотири консервативних залишки цистеїну, а в комплексі I бактерії Aquifex aeolicus виявили додатковий Fe4S4 кластер, що з'єднує N7 із рештою кластерів. Із цього випливає висновок, що в A. aeolicus комплекс I, окрім НАДН, може використовувати інші донори електронів, які б передавали останніх через N7[20].
Збірка мітохондріального комплексу I
У ссавців у процесі збірки 44 субодиниць НАДН-дегідрогеназного комплексу беруть участь 13 факторів збірки. Правильне функціонування та координація дії цих факторів прямим чином впливають на процес збірки комплексу, що своєю чергою впливає на правильне функціонування дихального ланцюга мітохондрій та сигнальні системи клітини.
Наразі побутує складна модулярна модель збірки комплексу. Відповідно до неї, окремі молекули збираються незалежно одна від іншої, а відтак відбувається міжмолекулярна асоціація з утворенням ферменту[21].
Мітохондріальний комплекс I утворює з дихальними комплексами III і IV суперкомплекси, так звані респірасоми. У мітохондріях ссавців і людини близько 90 % комплексу перебуває в складі респірасом. Також на мітохондріях із молодих кореневищ бамбука виявлено, що 90 % загальної кількості комплексу I зібрано в распірасоми, а в Різушки Таля — у суперкомплекс I—III2[22]. Існує достатньо доказів того, що присутність респірасом є необхідною умовою для стабільності та функціонування комплексу I, який за відсутності комплексів III чи IV є нестабільним. Наприклад, на мутантних клітинах людини виявлено, що комплекс I є необхідним для формування комплексу III, і навпаки, втрата комплексу III призводить до втрати комплексу I. Окрім того, у низці досліджень на тваринних клітинах наводяться докази того, що для стабільності комплексу I є необхідні комплекси IV і димер комплексу III.
У 2012 р. на культурі клітин людини виявили, що комплекси III і IV є необхідні для збірки повноцінонного комплексу I, при цьому сам неповністю зібраний комплекс слугує основою для утворення респірасом. Наявність у респірасомі комплексів III і IV є обов'язковою умовою для приєднання до комплексу I каталітичних субодиниць НАДН-дегідрогеназного модуля, які повністю активують комплекс і всю респірасому[23].
Реакція
НАДН-дегідрогеназний комплекс окиснює НАДН, що утворився в матриксі протягом циклу Кребса. Електрони з НАДН використовуються для відновлення мембранного переносника, убіхінону Q, який транспортує їх до наступного комплексу електрон-транспортного ланцюга мітохондрій, комплексу III або цитохром-bc1-комплексу[24].
НАДН-дегідрогеназний комплекс працює як протонна помпа: на кожен окиснений НАДН і відновлений Q через мембрану в міжмембранний простір перекачуються чотири протони[25]:
- НАДН + H+ + Q + 4H+in → НАД+ + QH2 + 4H+out
Утворений під час реакції електрохімічний потенціал використовується для синтезу АТФ. Цікаво, що реакція, що каталізується комплексом, буває оберненою, цей процес називається аеробне сукцинат-індуковане відновлення НАД+. В умовах високого потенціалу на мембрані та надлишку відновлених убіхінолів комплекс може відновлювати НАД+ із використанням їх електронів і пропускати протони назад у матрикс. Цей феномен зазвичай спостерігають при великих кількостях сукцинату і малих оксалоацетату та малату. Відновлення убіхінону здійснюється ферментами сукцинатдегідрогеназою, гліцерол-3-фосфатдегідрогеназою або мітохондріальною дегідрооротатдегідрогеназою. В умовах високого протонного градієнту спорідненість комплексу до убіхінолу підвищується, а редокс-потенціал убіхінолу падає через підвищення його концентрації, що й робить можливим зворотний транспорт електронів за електричним потенціалом внутрішньої мембрани мітохондрії до НАД[26]. Цей феномен вдалось спостерігати в лабораторних умовах, але залишається невідомим, чи проявляється він у живій клітині.
Механізм транспорту протонів

На початкових етапах дослідження комплексу I жваво обговорювали модель, що ґрунтується на припущені, що в комплексі оперує система, схожа з Q-циклом. Однак пізніші дослідження не виявили в комплексі I яких-небудь внутрішньо зв'язаних хінонів і повністю спростували цю гіпотезу[27]. НАДН-дегідрогеназний комплекс, імовірно, має унікальний механізм транспорту протонів шляхом конформаційних змін самого ферменту. Субодиниці ND2, ND4 та ND5 називають антипорт-подібними, оскільки вони є гомологічними один для одного і для бактеріальних Mrp Na+/H+ антипортів. Ці три субодиниці утворюють три основних протонних канали, що складаються з консервативних залишків заряджених амінокислот (в основному лізину та глутамату). Четвертий протонний канал утворений частиною субодиниці Nqo8 і малими субодиницями ND6, ND4L і ND3. Канал схожий за будовою з гомологічними каналами антипорт-схожих субодиниць, але містить незвичайно велику кількість густо упакованих залишків глутамату з боку матриксу, за що й отримав назву E-канал (латинське E використовують за звичайне позначення глутамату). Від C-кінця субодиниці ND5 відходить продовження, що складається з двох трансмембранних α-спіралей, об'єднаних незвичайно довгою (110 Å) α-спіраллю[6] (HL), яка, проходячи з боку комплексу, зануреного в матрикс, фізично сполучає всі антипорт-схожі субодиниці, і можливо, бере участь у спряженні транспорту електронів із конформаційною перебудовою. Ще один елемент спряження, βH, утворений серією β-шпильок, що перекриваються, та α-спіралей, він міститься на протилежному, периплазматичному, боці комплексу[28].
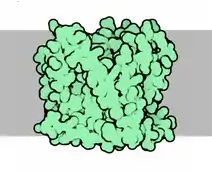
До сьогодні остаточно невідомо, як саме транспорт електронів спряжений із перенесенням протонів. Припускають, що потужний негативний заряд кластера N2 може розштовхувати навколишні поліпептиди, викликаючи цим конформаційні зміни, які певним чином поширюються на антипорт-схожі субодиниці, що розміщені досить далеко одна від одної. Відповідно до іншої гіпотези, унаслідок зміни конформації, в незвичайно довгому сайті зв'язування убіхінону утворюється стабілізований убіхінол Q−2 з вкрай низьким редокс-потенціалом і негативним зарядом. Невідомими залишаються і багато деталей щодо кінетики конформаційних змін і спряженого з ними транспорту протонів[28].
Активна та неактивна форми
Еукаріотичний НАДН-дегідрогеназний комплекс існує в двох чітко розрізнюваних формах: одна є повноцінно функціональною, так звана неактивна або D-форма. Якщо фермент перебуває при підвищених, але все ще оптимальних температурах (> 30 °C) за відсутності субстрату, ензим переходить у D-форму. Вона є каталітично неактивною, але може активуватись субстратом (НАДН та убіхіноном, на який можна скидати електрони). Після одного чи декількох ферментативних циклів комплекс стає активним, а швидкість реакції підвищується. Такий перехід виявили лише в хребетних і грибів, на відміну від безхребетних і бактерій. Рослинні комплекси ще не вивчали. За присутності двовалентних катіонів (Mg2+, Ca2+) чи в лужному pH активація відбувається значно триваліше, а вільна пальмітинова кислота значно збільшує частоту переходу з активної форми в деактивовану[29].
 |  | |||||||||
Комплекс I з бика та гриба Yarrowia lipolytica | ||||||||||
Висока енергія активації (270 кДж/моль) переходу з A- в D-форму вказує на те, що в комплексі відбувається значна конформаційна перебудова. До сьогодні єдиною виявленою різницею між двома формами залишається кількість залишків цистеїну на поверхні ферменту. Відповідно до останніх даних, у цьому процесі беруть участь субодиниці, що містяться поруч із хінон-зв'язувальним сайтом: 39 кДа, ND3 та ND1[29]. Обробка D-форм комплексу I спеціальними реагентами (N-етилмалеїмідом або реактивом Еллмана) незворотньо блокує ці важливі залишки цистеїну, роблячи цим самим неможливою повторну активацію ферменту. Цікаво, що A-форма комплексу I є не чутливою до тіолів, що вказує на те, що залишки цистеїну заховані глибоко в білку. Своєю чергою, деактивована форма є чутливою до інгібування нітрозотріолами та пероксинітритом[30].
Конформаційні зміни комплексу I мають велике фізіологічне значення. Після гіпоксії відновлення рівня кисню може призвести до спалаху окиснення НАД(Ф)Н і генерації активних форм кисню (АФК), які можуть пошкодити мітохондрії та викликати некроз тканин. Перехід із активної в неактивну форму комплексу відбувається за патологічних станів, коли кількість обертів ферменту є зниженою за нормальної, фізіологічної температури тіла, наприклад, при гіпоксії, ішемії чи підвищенні кількості оксиду азоту (NO) в тканинах (так звана метаболічна гіпоксія). Таким чином комплекс I запобігає окисненню решти дихальних комплексів, коли відбувається відновлення рівня кисню. Окрім цього, неактивна форма є нездатною до зворотного транспорту електронів, що знижує утворення АФК[29][31].
Еволюційне походження

НАДН-дегідрогеназний комплекс відносять до родини мембранних оксидоредуктаз із класу NiFe-гідрогеназ, які в анаеробних бактерій та архей спрягають реакцію окиснення субстрату та відновлення водню з транспортом протонів. Відштовхуючись від даних про гомологію білків, можна зробити висновок, що комплекс виник унаслідок сполучення двох комплексів, що вже існували, з різних неспоріднених білкових родин. НАДН-дегідрогеназний та убіхінон-зв'язувальний модулі виникли з розчинної NiFe-гідрогенази, яка окиснювала НАДН і відновлювала водень, а гідрофобна мембранна «підошва» комплексу, що перекачує протони, виникла з Na+/H+-антипортів Mrp[6].

Зливання розчинної гідрогенази та білків-антипортів призвело до виникнення великої кількості мембранних гідрогеназ і дегідрогеназ, які могли відтак еволюціонувати в комплекс I. Тривимірна структура цих ферментів, вірогідно, є схожою з такою в комплексі I. До дегідрогеназ можна віднести архейний комплекс Fpo з 11 субодиниць, що окиснює кофактор F420, що є зв'язаним із воднем, і відновлює метанофеназин (аналог убіхінону), перекачуючи через мембрану один протон на два електрони. У цього ферменту немає НАДН-дегідрогеназного модуля. До групи гідрогеназ відносять форміатгідрогенліази з кишкової палички (лат. Escherichia coli): форміатгідрогенліазу-1 із семи субодиниць і форміатгідрогенліазу-2 з десяти. Обидва ферменти окиснюють форміат, відновлюючи водень із перенесенням декількох протонів через мембрану[20].
Найпростішим із білків-родичів комплексу I є гідрогеназа Ech (англ. E. coli hydrogenase-3-type hydrogenase) археї Methanosarcina barkeri. Вона складається всього з шести субодиниць і перекачує один протон унаслідок окиснення фередоксину з відновленням молекули водню. Ech містить мінімальний набір субодиниць (гомологічних комплексу I), що є необхідним для спряження реакції окиснення з транспортом протонів[20].
Окрім цього, комплекс I виявили в хлоропластах у вигляді НАДФН-дегідрогеназного комплексу хлоропластів. Його будова та функція залишаються невідомими[32].
Утворення активних форм кисню
Комплекс I у процесі своєї роботи утворює активні форми кисню (АФК)[33]. Зазвичай це супероксид (а також пероксид гідрогену), і утворюється він щонайменш двома шляхами. У ході прямого транспорту електронів, у процесі дихання, утворюється дуже мала кількість супероксиду (вірогідно, менше 0.1 % усього потоку електронів переносяться на кисень)[34].
Під час зворотного транспорту електронів, який відбувається в умовах аеробного сукцинат-індукованого відновлення НАД+, комплекс I, можливо, стає найактивнішим місцем утворення супероксиду: до 5 % електронів ідуть на відновлення кисню[35].
Супероксид утворюється в НАДН-дегідрогеназному комплексі внаслідок перенесення одного електрону з ФМНН2 на O2. Утворений радикал флавіну є нестабільним і переносить електрон, що залишився, на залізосіркові кластери. Рівень утворення супероксиду визначається відношенням НАДН/НАД+; в умовах, коли відновлено невелику кількість НАД, НАД+ успішно конкурує за електрони з киснем[36][37].
Інгібітори
Найкраще вивченим інгібітором комплексу I є ротенон, що широко використовується як органічний пестицид. Ротенон і ротеноїди — це ізофлавоноїди, які є присутні в коренях деяких родів тропічних рослин, таких як Антонія (Loganiaceae), Derris і Lonchocarpus (Fabaceae). Ротенон давно використовується як інсектицид і рибна отрута, оскільки мітохондрії комах і риб є до нього особливо чутливими. Відомо, що корінні жителі Французької Гвіани та інші індіанці Південної Америки використовували ротенонвмісні рослини для риболовлі вже у XVII столітті[38]. Ротенон взаємодіє із сайтом зв'язування убіхінону та конкурує з основним субстратом. Доведено, що тривале системне пригнічування комплексу I ротеноном здатне індукувати селективне відмирання дофамінергічних нейронів (тих, що секретують нейротрансмітер дофамін)[39]. Подібно діє і пієрицидин А, ще один потужний інгібітор комплексу I, структурно подібний до убіхінону. До цієї ж групи відносять і амітал натрію — похідну барбітурової кислоти[4].
Незважаючи на понад 50-річне вивчення комплексу I, так і не вдалось виявити інгібітори, які блокували б перенесення електронів у межах комплексу. Гідрофобні інгібітори, такі як ротенон чи пієрицидин, просто переривають перенесення електрона з термінального кластера N2 на убіхінон.
Ще одна речовина, що блокує комплекс I — це аденозиндифосфатрибоза, конкурентний інгібітор у реакції окиснення НАДН. Вона зв'язується з ферментом у сайті зв'язування нуклеотиду (ФАД)[40].
До найпотужніших інгібіторів комплексу I відносять родину ацетогенінів. Виявлено, що ці речовини утворюють хімічні зшивання із субодиницею ND2, що опосередковано вказує на роль ND2 у зв'язуванні убіхінону[41]. Цікаво, що ацетогенін роллініастатин-2 став першим із виявлених інгібіторів комплексу I, які зв'язуються у відмінному від ротенону місці[42].
Помірним інгібіторним ефектом володіє протидіабетний препарат метформін; очевидно, дана властивість препарату закладена в основі механізму його дії[43].
Патології
Мутації в генах субодиниць комплексу I можуть викликати мітохондріальні захворювання, наприклад, синдром Лея. Точкові мутації мітохондріальних субодиниць цього комплексу також можуть спричинити спадкову зорову нейропатію Лебера. Існують докази, що дефекти в структурі комплексу I можуть відігравати роль в етіології хвороби Паркінсона, можливо, через утворення активних форм кисню[44]. Так, було виявлено, що в культур клітин хворих хворобою Паркінсона посилений відтік протонів у комплексі I, що знижує максимальну дихальну ємність легень[45]. У рослин мутації генів, що кодують поліпептидні ланцюги комплексу I, описані в тютюну (Nicotiana silvestris) та кукурудзи (Zea mays): мутації супроводжувались патологією пилку та призводили до цитоплазматичної стерильності в чоловіків[4].
Дослідження 2010 р. виявили незвичайну роль комплексу I у роботі мозку. Активність цього ферменту значно знижена в пацієнтів із біполярним розладом, але залишається нормальною в пацієнтів із депресією чи шизофренією. У пацієнтів із біполярним розладом у префронтальній корі спостерігалось посилене окиснення та нітрування білків. Ці результати роблять комплекс I мішенню для майбутніх терапевтичних досліджень біполярного розладу[46][47].
Дія пестицидів, що блокують комплекс I, може призвести до довготривалих наслідків. Наприклад, тривала дія низьких концентрацій органофосфату та пестициду дихлофосу викликає дисфункцію печінки. Дихлофос змінює активність комплексів I і II, що призводить до сповільнення транспорту електронів і зниження синтезу АТФ[48].
Роль комплексу I у процесах старіння
Дані чисельних досліджень свідчать про те, що мітохондрії, і зокрема комплекси I і II, відіграють ключову роль у процесах, що впливають на старіння та на тривалість життя[49][50][51][52]. Вважається, що сповільнення при старінні синтезу та відновлення білків призводить до збою в стехіометрії дихальних субодиниць. Це, своєю чергою, викликає порушення ефективності функціонування комплексу I і посилення мітохондріального окисного стресу, що найяскравіше виражено в м'язовій тканині[53].
Вставлення у додаток до комплексу I у геном дрозофіли альтернативної НАДH-дегідрогенази Ndi1 дріжджів, що складається всього з однієї субодиниці, призводило до відновлення нормального рівня окиснення внутрішньомітохондріального НАДH і значного збільшення тривалості життя цієї мухи незалежно від обмеження калорійності її дієти[54].
Див. також
- Цитохром-b6f-комплекс
- Цитохром-bc1-комплекс
- Альтернативна оксидаза
- ETF-дегідрогеназа
- Оксидоредуктази
- Кофактор (біохімія)
Примітки
- Губський, 2000, с. 127.
- Гонський, 2002, с. 266.
- Rouslan G. Efremov, Rozbeh Baradaran & Leonid A. Sazanov (27 травня 2010). The architecture of respiratory complex I. nature 465: 441–445. PMID 20505720. doi:10.1038/nature09066.
- Ермаков, 2005, с. 237.
- Carroll J, Fearnley IM, Skehel JM, Shannon RJ, Hirst J, Walker JE (October 2006). Bovine complex I is a complex of 45 different subunits. J. Biol. Chem. 281 (43): 32724–7. PMID 16950771. doi:10.1074/jbc.M607135200.
- Leonid A. Sazanov (2015 Jun). A giant molecular proton pump: structure and mechanism of respiratory complex I. Nature Reviews Molecular Cell Biology 16 (6): 375–388. PMID 25991374. doi:10.1038/nrm3997.(англ.)
- Judy Hirst (June 2013). Mitochondrial Complex I. Annual Review of Biochemistry 82: 551–575. doi:10.1146/annurev-biochem-070511-103700.
- Cardol P, Lapaille M, Minet P, Franck F, Matagne RF, Remacle C. (2006 Sep). ND3 and ND4L subunits of mitochondrial complex I, both nucleus encoded in Chlamydomonas reinhardtii, are required for activity and assembly of the enzym. Eukaryot Cell. 5 (9): 1460–7. PMID 16963630.
- Voet, Judith G.; Voet, Donald (2004). Biochemistry (вид. 3rd). New York: J. Wiley & Sons. с. 813–826. ISBN 0-471-19350-X.
- Balsa E, Marco R, Perales-Clemente E, Szklarczyk R, Calvo E, Landázuri MO, Enríquez JA (September 2012). NDUFA4 is a subunit of complex IV of the mammalian electron transport chain. Cell Metab. 16 (3): 378–86. PMID 22902835. doi:10.1016/j.cmet.2012.07.015.
- Allan G Rasmussonb, Volker Heiserc, Eduardo Zabaletaa, Axel Brennickea, Lutz Grohmannd (May 1998). Physiological, biochemical and molecular aspects of mitochondrial complex I in plants. Biochimica et Biophysica Acta (BBA) - Bioenergetics 1364 (2): 101–111. doi:10.1016/S0005-2728(98)00021-8.
- Peters K., Belt K., Braun H.-P. (2013). 3D gel map of Arabidopsis complex I. Front. Plant Sci. Plant Proteomics. 5 (153). PMID 23761796. doi:10.3389/fpls.2013.00153.
- Meyer E.H. (2012 May 24). Proteomic investigations of complex I composition: how to define a subunit?. Front. Plant Sci. Plant Proteomics. 3 (106). PMID 22654890. doi:10.3389/fpls.2012.00106.
- Cardol P (2011). Mitochondrial NADH:ubiquinone oxidoreductase (complex I) in eukaryotes: a highly conserved subunit composition highlighted by mining of protein databases.. Biochim Biophys Acta 1807 (11): 1390–7. PMID 21749854. doi:10.1016/j.bbabio.2011.06.015.
- Marion Babot, Amanda Birch, Paola Labarbuta, Alexander Galkin (July 2014). Characterisation of the active/de-active transition of mitochondrial complex I. Biochimica et Biophysica Acta (BBA) - Bioenergetics 1837 (7): 1083–1092. PMID 24569053. doi:10.1016/j.bbabio.2014.02.018.
- Katarzyna Kmita, Volker Zickermann (Oct 01, 2013,). Accessory subunits of mitochondrial complex I. Biochemical Society Transactions 41 (5): 1272–1279. doi:10.1042/BST20130091.
- Ogilvie I, Kennaway NG, Shoubridge EA (2005). A molecular chaperone for complex I assembly is mutated in a progressive encephalopathy.. J Clin Invest 115 (10): 2784–92. PMC 1236688. PMID 16200211. doi:10.1172/JCI26020.
- Dunning CJ, McKenzie M, Sugiana C, Lazarou M, Silke J, Connelly A, etal (2007). Human CIA30 is involved in the early assembly of complex I and mutations in its gene cause disease.. EMBO J 26 (13): 3227–37. PMC 1914096. PMID 17557076. doi:10.1038/sj.emboj.7601748.
- Saada A, Vogel RO, Hoefs SJ, van den Brand MA, Wessels HJ, Willems PH, etal (2009). Mutations in NDUFAF3 (C3ORF60), encoding an NDUFAF4 (C6ORF66)-interacting complex I assembly protein, cause fatal neonatal disease.. Am J Hum Genet 84 (6): 718–27. PMC 2694978. PMID 19463981. doi:10.1016/j.ajhg.2009.04.020.
- Rouslan G. Efremov, Leonid A. Sazanov (October 2012). The coupling mechanism of respiratory complex I — A structural and evolutionary perspective. Biochimica et Biophysica Acta 1817 (10): 1785–1795. PMID 22386882. doi:10.1016/j.bbabio.2012.02.015.
- Sánchez-Caballero, Laura; Guerrero-Castillo, Sergio; Nijtmans, Leo (1 липня 2016). Unraveling the complexity of mitochondrial complex I assembly: A dynamic process. Biochimica Et Biophysica Acta 1857 (7). с. 980–990. ISSN 0006-3002. PMID 27040506. doi:10.1016/j.bbabio.2016.03.031. Процитовано 9 червня 2016.
- Eubel H., Jänsch L., Braun H.P. (2003). New insights into the respiratory chain of plant mitochondria: supercomplexes and a unique composition of complex II. Plant Physiol. 133: 274–286. PMID 12970493. doi:10.1104/pp.103.024620.
- David Moreno-Lastres, Flavia Fontanesi, Inés García-Consuegra, Miguel A. Martín, Joaquín Arenas, Antoni Barrientos, and Cristina Ugalde1 (Mar 7). Mitochondrial Complex I plays an Essential Role in Human Respirasome Assembly. Cell Metab. 15 (3): 324–335. doi:10.1016/j.cmet.2012.01.015.
- Berg, J, Tymoczko, J, and L Stryer (2006). Biochemistry (вид. 6th). New York: WH Freeman & Company. с. 509–513.
- Brandt, U (2006). Energy converting NADH:quinone oxidoreductase (complex I). Annual Review of Biochemistry 75: 69–92. PMID 16756485. doi:10.1146/annurev.biochem.75.103004.142539.
- Grivennikova VG, Kotlyar AB, Karliner JS, Cecchini G, Vinogradov AD. (August 2007). Redox-dependent change of nucleotide affinity to the active site of the mammalian complex I.. Biochemistry 46 (38): 10971–8. PMC 2258335. PMID 17760425. doi:10.1021/bi7009822.
- Ермаков, 2005, с. 238.
- Rozbeh Baradaran, John M. Berrisford, Gurdeep S. Minhas & Leonid A. Sazanov (28 лютого 2013). Crystal structure of the entire respiratory complex I. Nature 494: 443–448. doi:10.1038/nature11871.
- Marion Babot, Amanda Birch, Paola Labarbuta, Alexander Galkin (July 2014). Characterisation of the active/de-active transition of mitochondrial complex I. Biochimica et Biophysica Acta (BBA) - Bioenergetics 1837 (7): 1083–1092. doi:10.1016/j.bbabio.2014.02.018.
- Galkin A, Moncada S (December 2007). S-nitrosation of mitochondrial complex I depends on its structural conformation. J. Biol. Chem. 282 (52): 37448–53. PMID 17956863. doi:10.1074/jbc.M707543200.
- Moncada S, Erusalimsky JD (March 2002). Does nitric oxide modulate mitochondrial energy generation and apoptosis?. Nat. Rev. Mol. Cell Biol. 3 (3): 214–20. PMID 11994742. doi:10.1038/nrm762.
- Lianwei Peng, Hiroshi Yamamoto, Toshiharu Shikanai (August 2011). Structure and biogenesis of the chloroplast NAD(P)H dehydrogenase complex. Biochimica et Biophysica Acta 1807 (8): 945–953. doi:10.1016/j.bbabio.2010.10.015.
- Murphy MP (January 2009). How mitochondria produce reactive oxygen species. Biochem. J. 417 (1): 1–13. PMC 2605959. PMID 19061483. doi:10.1042/BJ20081386.
- Hansford RG, Hogue BA, Mildaziene V (February 1997). Dependence of H2O2 formation by rat heart mitochondria on substrate availability and donor age. J. Bioenerg. Biomembr. 29 (1): 89–95. PMID 9067806. doi:10.1023/A:1022420007908.
- Muller FL, Liu Y, Abdul-Ghani MA, Lustgarten MS, Bhattacharya A, Jang YC, Van Remmen H (January 2008). High rates of superoxide production in skeletal-muscle mitochondria respiring on both complex I- and complex II-linked substrates. Biochem. J. 409 (2): 491–9. PMID 17916065. doi:10.1042/BJ20071162.
- Kussmaul L, Hirst J. (May 2006). The mechanism of superoxide production by NADH:ubiquinone oxidoreductase (complex I) from bovine heart mitochondria. Proceedings of the National Academy of Sciences of the United States of America 103 (20): 7607–12. PMC 1472492. PMID 16682634. doi:10.1073/pnas.0510977103.
- Esterházy D, King MS, Yakovlev G, Hirst J. (March 2008). Production of reactive oxygen species by complex I (NADH:ubiquinone oxidoreductase) from Escherichia coli and comparison to the enzyme from mitochondria. Biochemistry 25 (12): 3964–71. PMID 18307315. doi:10.1021/bi702243b.
- Moretti C, Grenand P (September 1988). [The "nivrées", or ichthyotoxic plants of French Guyana]. J Ethnopharmacol (French) 6 (2): 139–60. PMID 7132401. doi:10.1016/0378-8741(82)90002-2.
- Watabe M, Nakaki T. (July 2008). Mitochondrial complex I inhibitor rotenone inhibits and redistributes vesicular monoamine transporter 2 via nitration in human dopaminergic SH-SY5Y cells. Molecular Pharmocology 74 (4): 933–40. PMID 18599602. doi:10.1124/mol.108.048546.
- Zharova TV, Vinogradov AD. (July 1997). A competitive inhibition of the mitochondrial NADH-ubiquinone oxidoreductase (complex I) by ADP-ribose. Biochimica et Biophysica Acta 1320 (3): 256–64. PMID 9230920. doi:10.1016/S0005-2728(97)00029-7.
- Nakamaru-Ogiso E, Han H, Matsuno-Yagi A, Keinan E, Sinha SC, Yagi T, Ohnishi T. (January 2010). The ND2 subunit is labeled by a photoaffinity analogue of asimicin, a potent complex I inhibitor.. FEBS Letters 584 (5): 883–8. PMC 2836797. PMID 20074573. doi:10.1016/j.febslet.2010.01.004.
- Degli Esposti M, Ghelli A, Ratta M, Cortes D, Estornell E. (July 1994). Natural substances (acetogenins) from the family Annonaceae are powerful inhibitors of mitochondrial NADH dehydrogenase (complex I). The Biochemical Journal 301: 161–7. PMC 1137156. PMID 8037664.
- Viollet B, Guigas B, Sanz Garcia N, Leclerc J, Foretz M, Andreelli F. (March 2012). Cellular and molecular mechanisms of metformin: an overview. Clinical Science (London) 122 (6): 253–70. PMID 22117616. doi:10.1042/CS20110386.
- Chou AP, Li S, Fitzmaurice AG, Bronstein JM. (April 2010). Mechanisms of rotenone-induced proteasome inhibition. Neurotoxicology 113 (4): 674–82. PMC 2885979. PMID 20417232. doi:10.1016/j.neuro.2010.04.006.
- Esteves AR, Lu J, Rodova M, Onyango I, Lezi E, Dubinsky R, Lyons KE, Pahwa R, Burns JM, Cardoso SM, Swerdlow RH. (February 2010). Mitochondrial respiration and respiration-associated proteins in cell lines created through Parkinson's subject mitochondrial transfer. Journal of Neurochemistry 113 (3): 674–82. PMID 20132468. doi:10.1111/j.1471-4159.2010.06631.x.
- Andreazza AC, Shao L, Wang JF, Young LT. (April 2010). Mitochondrial complex I activity and oxidative damage to mitochondrial proteins in the prefrontal cortex of patients with bipolar disorder. Archives of General Psychiatry 67 (4): 360–8. PMID 20368511. doi:10.1001/archgenpsychiatry.2010.22.
- Moran M, Rivera H, Sánchez-Aragó M, Blázquez A, Merinero B, Ugalde C, Arenas J, Cuezva JM, Martín MA. (May 2010). Mitochondrial bioenergetics and dynamics interplay in complex I-deficient fibroblasts. Biochimica et Biophysica Acta 1802 (5): 443–53. PMID 20153825. doi:10.1016/j.bbadis.2010.02.001.
- Binukumar BK, Bal A, Kandimalla R, Sunkaria A, Gill KD. (April 2010). Mitochondrial energy metabolism impairment and liver dysfunction following chronic exposure to dichlorvos. Toxicology 270 (2–3): 77–84. PMID 20132858. doi:10.1016/j.tox.2010.01.017.
- Stefanatos, R., & Sanz, A. (2011). Mitochondrial complex I: a central regulator of the aging process. Cell Cycle, 10(10), 1528—1532
- Scialo, F., Mallikarjun, V., Stefanatos, R., & Sanz, A. (2013). Regulation of lifespan by the mitochondrial electron transport chain: reactive oxygen species-dependent and reactive oxygen species-independent mechanisms. Antioxidants & redox signaling, 19(16), 1953—1969. DOI:10.1089/ars.2012.4900
- López-Lluch, G., Santos-Ocaña, C., Sánchez-Alcázar, J. A., Fernández-Ayala, D. J. M., Asencio-Salcedo, C., Rodríguez-Aguilera, J. C., & Navas, P. (2015). Mitochondrial responsibility in ageing process: innocent, suspect or guilty. Biogerontology, 16(5), 599—620. DOI:10.1007/s10522-015-9585-9
- Bowman, A., & Birch-Machin, M. A. (2016). The age-dependent decrease of mitochondrial complex II activity in human skin fibroblasts. Journal of Investigative Dermatology. doi:10.1016/j.jid.2016.01.017
- Kruse, S. E., Karunadharma, P. P., Basisty, N., Johnson, R., Beyer, R. P., MacCoss, M. J., Rabinovitch, P. S. and Marcinek, D. J. (2016), Age modifies respiratory complex I and protein homeostasis in a muscle type-specific manner. Aging Cell, . Aging cell, 15(1), 89-99. DOI:10.1111/acel.12412
- Sanz, A., Soikkeli, M., Portero-Otín, M., Wilson, A., Kemppainen, E., McIlroy, G., … & Kiviranta, E. (2010). Expression of the yeast NADH dehydrogenase Ndi1 in Drosophila confers increased lifespan independently of dietary restriction. Proceedings of the National Academy of Sciences, 107(20), 9105-9110. DOI:10.1073/pnas.0911539107 Повний текст на PMC: 2889079
Література
Українською
- Губський Ю. Біологічна хімія. — Київ-Тернопіль : Укрмедкнига, 2000. — 508 с. — ISBN 966-7364-41-0.
- Гонський Я., Максимчук Т., Калинський М. Біохімія людини. — Тернопіль : Укрмедкнига, 2002. — 744 с. — ISBN 966-7364-17-8.
Англійською
- Judy Hirst. Mitochondrial Complex I. Annual Review of Biochemistry. — 2013. — Vol. 82. — P. 551–575. — DOI:
- Leonid A. Sazanov. A giant molecular proton pump: structure and mechanism of respiratory complex I. Nature Reviews Molecular Cell Biology. — 2015. — Vol. 16. — P. 375–388. — DOI: — PMID:
- Carroll J, Fearnley IM, Skehel JM, Shannon RJ, Hirst J, Walker JE. Bovine complex I is a complex of 45 different subunits. — 2006. — Vol. 281. — DOI: — PMID:
- Voet, Judith G.; Voet, Donald. Biochemistry. — New York : J. Wiley & Sons, 2004. — P. 813–826. — ISBN 0-471-19350-X.
- Marion Babot, Amanda Birch, Paola Labarbuta, Alexander Galkin. Characterisation of the active/de-active transition of mitochondrial complex I. — Biochimica et Biophysica Acta (BBA) - Bioenergetics, 2014. — Vol. 7. — P. 1083–1092. — DOI:
- Katarzyna Kmita, Volker Zickermann. Accessory subunits of mitochondrial complex I. — Biochemical Society Transactions, 2013. — Vol. 5. — P. 1272–1279. — DOI:
Російською
- Физиология растений / Під ред. И. П. Ермакова. — Москва : Академия, 2005. — 634 с.
Посилання
- MRC MBU Sazanov group
- Complex I home page at The Scripps Research Institute
- MeSH Electron+Transport+Complex+I